Análise econométrica em tempo real: Modelos família ARCH para dados financeiros
Rodrigo H. Ozon
21/09/2020
Resumo
Este artigo demonstra um case de aplicação dos modelos de heterocedasticidade condicional para cotações de ativos financeiros (em especial da VALE), com integração via scrapping direto do site do Yahoo!Finance. As cotações apresentam um delay de aproximadamente 1 dia de negociação, e os scripts dos chunks do R estão todos disponíveis para replicações futuras.
Palavras-Chave: ARCH, VALE, News Impact Curve, Econometria
Introdução
Esse relatório capta dinamicamente as cotações de tres ativos
PBRPetrobrasVALEVale^BVSPIndice Bovespa
Os dados são originários do Yahoo finance. As séries temporais se iniciam em 01/01/2018 e terminam no último valor de fechamento quase que em real time.
Navegue nele para ver a aplicação de modelos econométricos de volatilidade condicional para os dados da VALE.
Iniciamos observando o caso da maior estatal brasileira, a petrolífera Petrobras:
PBR Preço de Fechamento do pregão
PBR Volume negociado
A Operação Lava Jato é um conjunto de investigações, algumas controversas, em andamento pela Polícia Federal do Brasil, que cumpriu mais de mil mandados de busca e apreensão, de prisão temporária, de prisão preventiva e de condução coercitiva, visando apurar um esquema de lavagem de dinheiro que movimentou bilhões de reais em propina.
A operação teve início em 17 de março de 2014 e conta com 71 fases operacionais autorizadas, entre outros, pelo então juiz Sérgio Moro, durante as quais prenderam-se e condenaram-se mais de cem pessoas. Investiga crimes de corrupção ativa e passiva, gestão fraudulenta, lavagem de dinheiro, organização criminosa, obstrução da justiça, operação fraudulenta de câmbio e recebimento de vantagem indevida. A Lava Jato foi apontada por críticos como uma das causas da crise político-econômica de 2014 no país. De acordo com investigações e delações premiadas, estavam envolvidos em corrupção membros administrativos da empresa estatal petrolífera Petrobras, políticos dos maiores partidos do Brasil, incluindo presidentes da República, presidentes da Câmara dos Deputados e do Senado Federal e governadores de estados, além de empresários de grandes empresas brasileiras. A Polícia Federal considera-a a maior investigação de corrupção da história do país.
^BVSP Preço de Fechamento do pregão
^BVSP Volume negociado
O índice bovespa, também chamado de IBOV, é o principal indicador médio de desempenho do mercado de ações no Brasil.
É chamado o “termômetro do mercado de ações”. Se um investidor quer saber como está sua carteira de ações, ele compara com o desempenho do índice bovespa. Se ele está melhor, pior ou com desempenho similar ao do índice.
Formado pela composição de ações das empresas com maior liquidez e maior volume financeiro negociado de todo o volume de negócios da bolsa.
VALE Preço de Fechamento do pregão
VALE Volume negociado
O rompimento de barragem em Brumadinho em 25 de janeiro de 2019 foi o maior acidente de trabalho no Brasil em perda de vidas humanas e o segundo maior desastre industrial do século. Foi um dos maiores desastres ambientais da mineração do país, depois do rompimento de barragem em Mariana.
Controlada pela Vale S.A., a barragem de rejeitos denominada barragem da Mina Córrego do Feijão, era classificada como de “baixo risco” e “alto potencial de danos” pela empresa. Acumulando os rejeitos de um mina de ferro, ficava no ribeirão Ferro-Carvão, na região de Córrego do Feijão, no município de Brumadinho, estado de Minas Gerais.
Volatilidade Variável no Tempo e Modelos ARCH
O modelo autoregressivo de heterocedasticidade condicional (ARCH) diz respeito a séries temporais com heterocedasticidade variável no tempo, onde a variância é condicional à informação existente em um determinado ponto no tempo.
O modelo ARCH
O modelo ARCH assume que a média condicional do termo de erro em um modelo de série temporal é constante (zero), ao contrário da série não estacionária, mas sua variância condicional não. Esse modelo pode ser descrito como nas Equações 1, 2 e 3.
\[\begin{equation} y_{t}=\phi +e_{t} \label{eq:archdefA14} \end{equation}\]
\[\begin{equation} e_{t} | I_{t-1} \sim N(0,h_{t}) \label{eq:archdefB14} \end{equation}\]
\[\begin{equation} h_{t}=\alpha_{0}+\alpha_{1}e_{t-1}^2, \;\;\;\alpha_{0}>0, \;\; 0\leq \alpha_{1}<1 \label{eq:archdefC14} \end{equation}\]
As Equações 4 e 5 fornecem o modelo de teste e as hipóteses para testar os efeitos ARCH em uma série de tempo, onde os resíduos e \(\hat e_{t}\) vêm da regressão da variável \(y_{t}\) em uma constante, como 1, ou em uma constante mais outros regressores; o teste mostrado na Equação 4 pode incluir vários termos de defasagem, caso em que a hipótese nula (Equação 5) seria que todos eles são conjuntamente insignificantes.
\[\begin{equation} \hat e_{t}^2 = \gamma_{0}+\gamma_{1}\hat e_{t-1}^2+...+\gamma_{q}e_{t-q}^2+\nu_{t} \label{eq:archeffectseqA14} \end{equation}\]
\[\begin{equation} H_{0}:\gamma_{1}=...=\gamma_{q}=0\;\;\;H_{A}:\gamma_{1}\neq 0\;or\;...\gamma_{q}\neq 0 \label{eq:archeffectseqB14} \end{equation}\]
A hipótese nula é que não há efeitos ARCH. A estatística de teste é
\[ (T-q)R^2 \sim \chi ^2_{(1-\alpha,q)} \]
Rodando o modelo ARCH e GARCH para os dados da VALE
ativo2[1:10,] # Gero as primeiras 10 linhas do dataset VALE.Open VALE.High VALE.Low VALE.Close VALE.Volume VALE.Adjusted
2018-01-02 12.55 12.80 12.51 12.77 19189400 9.018118
2018-01-03 12.80 12.87 12.68 12.85 20449600 9.074613
2018-01-04 13.03 13.09 12.82 12.83 22033100 9.060488
2018-01-05 12.80 13.09 12.73 13.09 20042800 9.244100
2018-01-08 13.26 13.32 13.18 13.32 17507000 9.406524
2018-01-09 13.40 13.42 13.18 13.24 31204900 9.350030
2018-01-10 13.10 13.21 13.05 13.15 16253500 9.286470
2018-01-11 13.21 13.46 13.20 13.45 15011400 9.498329
2018-01-12 13.51 13.57 13.42 13.53 19012500 9.554827
2018-01-16 13.28 13.31 13.08 13.17 40821800 9.300596Cl(ativo2) # Gero os dados para aqueles que desejem baixar por aqui VALE.Close
2018-01-02 12.77
2018-01-03 12.85
2018-01-04 12.83
2018-01-05 13.09
2018-01-08 13.32
2018-01-09 13.24
2018-01-10 13.15
2018-01-11 13.45
2018-01-12 13.53
2018-01-16 13.17
...
2023-05-26 13.26
2023-05-30 12.76
2023-05-31 12.68
2023-06-01 13.04
2023-06-02 13.68
2023-06-05 13.61
2023-06-06 13.74
2023-06-07 13.94
2023-06-08 14.09
2023-06-09 14.04Vo(ativo2) # Gero os dados para aqueles que desejem baixar por aqui VALE.Volume
2018-01-02 19189400
2018-01-03 20449600
2018-01-04 22033100
2018-01-05 20042800
2018-01-08 17507000
2018-01-09 31204900
2018-01-10 16253500
2018-01-11 15011400
2018-01-12 19012500
2018-01-16 40821800
...
2023-05-26 19326400
2023-05-30 30416300
2023-05-31 45251700
2023-06-01 52730200
2023-06-02 29286200
2023-06-05 18834400
2023-06-06 23116100
2023-06-07 24257200
2023-06-08 15324000
2023-06-09 26761600# Voce pode usar o seguinte comando para exportar esse dataset
write.csv(ativo2, file="VALE.csv")
library(xlsx)
write.xlsx(ativo2, file="VALE.xlsx")
#https://www.guru99.com/r-exporting-data.html#1 Após vermos os precos de fechamento e volume, obtenho o log retorno dos precos
dlnVALEclose <- diff(log(Cl(ativo2)), lag=1) #ln(Pt/Pt-1)
dlnVALEclose<-dlnVALEclose[-1,]
presAnnotation <- function(dygraph, x, text) {
dygraph %>%
dyAnnotation(x, text, attachAtBottom = TRUE, width = 300, height=20)
}
dygraph(dlnVALEclose, main = "Retornos logarítmicos dos preços de fechamento VALE")%>%
dyAnnotation("2019-01-25", text = "A", tooltip = "Rompimento da barragem em Brumadinho")%>%
presAnnotation("2019-01-25", text = "Rompimento da barragem em Brumadinho")%>%
dyShading(from = "2019-01-25", to = "2019-02-26")%>%
dyAnnotation("2020-03-02", text = "A", tooltip = "Corona Day")%>%
presAnnotation("2020-03-02", text = "Corona Day")%>%
dyShading(from = "2020-03-02", to = "2020-04-27") #Grafico dos retornosAgora observo o padrão da distribuição dos log retornos:
hist(dlnVALEclose, main="", breaks=20, freq=FALSE, col="grey")
lines(density(dlnVALEclose, adjust=2, col="blue"))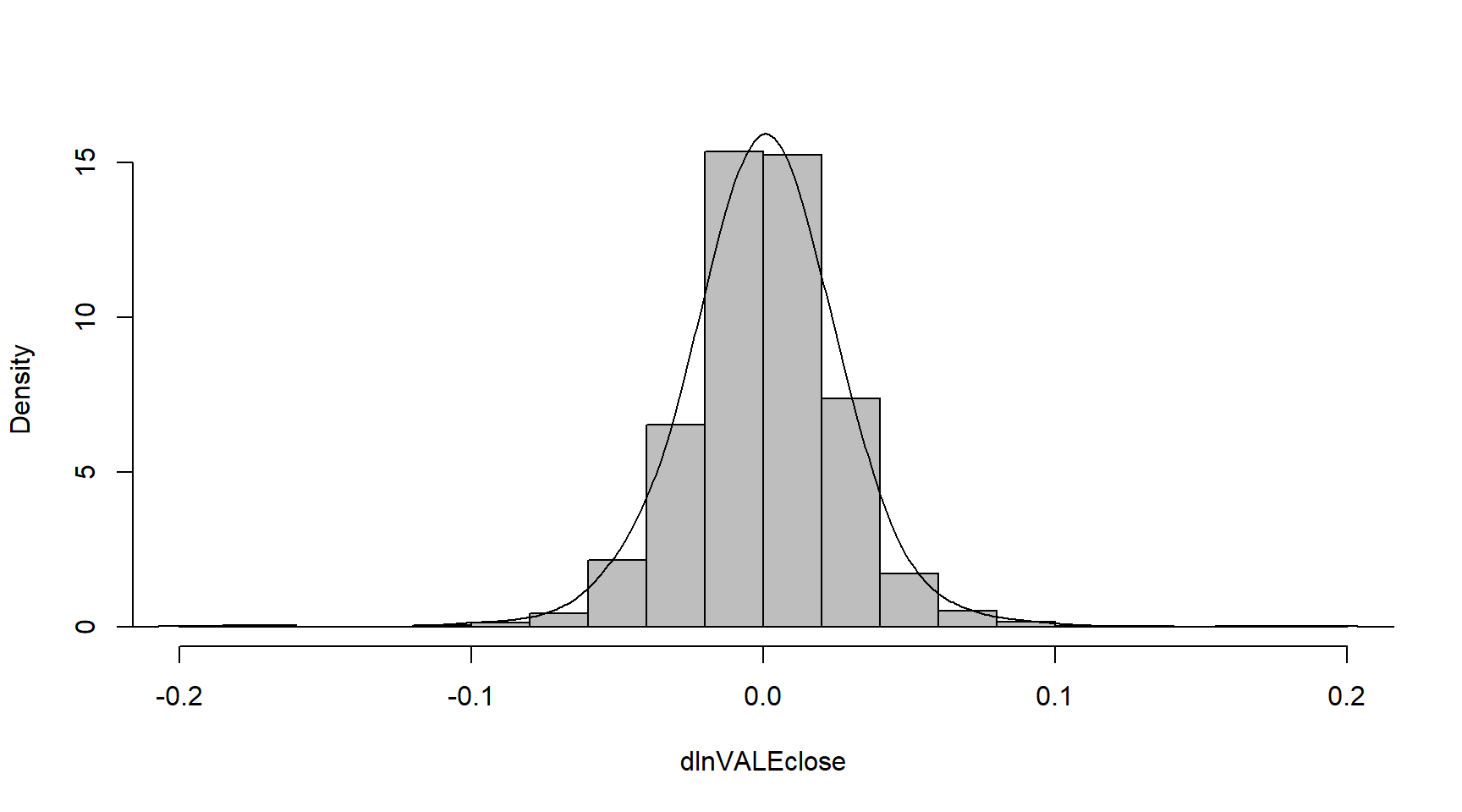
Vamos iniciar carregando os pacotes necessários do R:
#rm(list=ls()) #Removes all items in Environment!
library(FinTS) #for function `ArchTest()`
library(rugarch) #for GARCH models
library(tseries) # for `adf.test()`
library(dynlm) #for function `dynlm()`
library(vars) # for function `VAR()`
library(nlWaldTest) # for the `nlWaldtest()` function
library(lmtest) #for `coeftest()` and `bptest()`.
library(broom) #for `glance(`) and `tidy()`
#library(PoEdata) #for PoE4 datasets
library(car) #for `hccm()` robust standard errors
library(sandwich)
library(knitr) #for `kable()`
library(forecast) Vamos primeiro realizar, passo a passo, o teste ARCH na variável VALE.Close. Iremos transformar os dados numa série temporal para leitura do R:
library(dynlm)
rTS <- ts(dlnVALEclose) # A funcao ts eh o mesmo que timeseries
plot.ts(ativo2$VALE.Close) # Serie temporal de valor de fechamento da VALE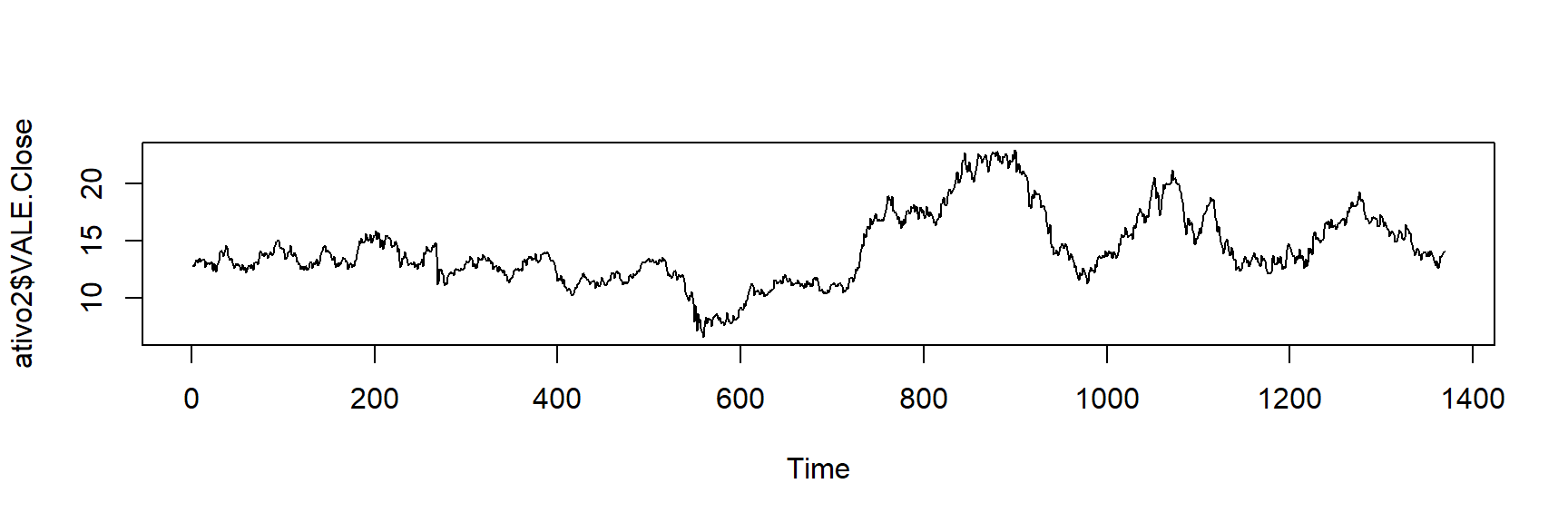
plot.ts(rTS) # Serie de retornos logaritmizados da VALE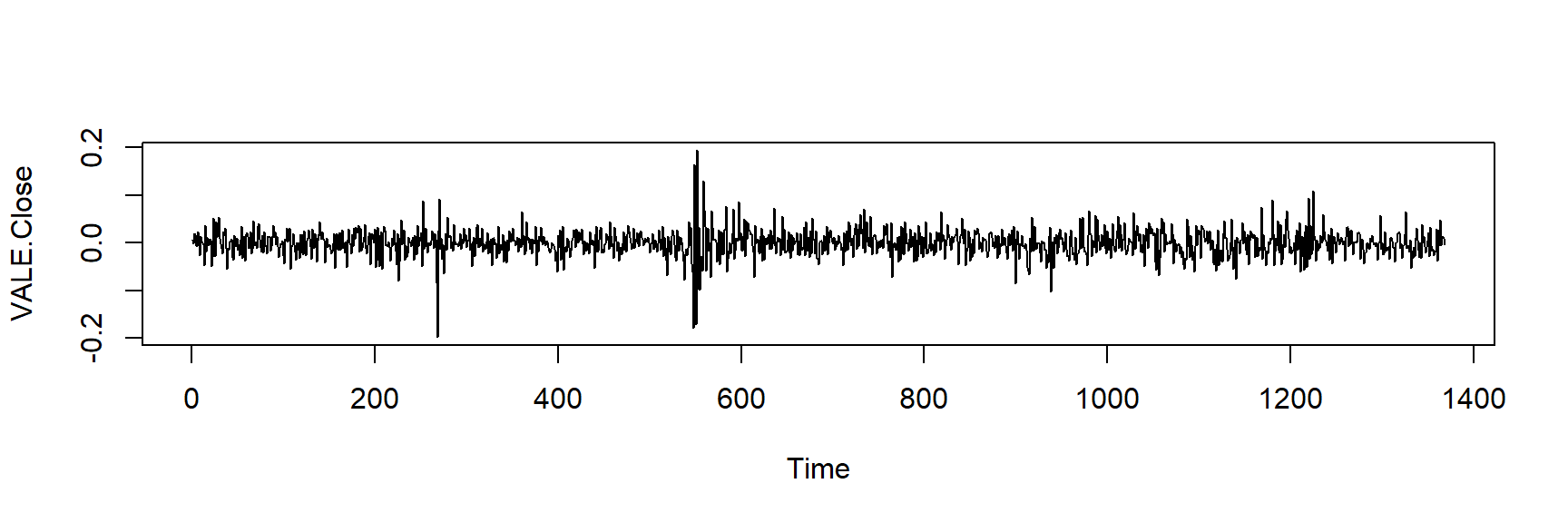
Então obtemos os valores de estimativas dos parâmetros da regressão:
VALE.media <- dynlm(rTS~1)
| Dependent variable: | |
| rTS | |
| Constant | 0.0001 |
| (0.001) | |
| Observations | 1,368 |
| R2 | 0.000 |
| Adjusted R2 | 0.000 |
| Note: | p<0.1; p<0.05; p<0.01 |
ehatsq <- ts(resid(VALE.media)^2) # Obtenho os valores da serie de residuos do modelo ARCH elevado ao quadrado
VALE.ARCH <- dynlm(ehatsq~L(ehatsq)) # Agora estimo o modelo dinamico usando a serie de residuos
summary(VALE.ARCH) # Printa os resultados
Time series regression with "ts" data:
Start = 2, End = 1368
Call:
dynlm(formula = ehatsq ~ L(ehatsq))
Residuals:
Min 1Q Median 3Q Max
-0.016531 -0.000548 -0.000380 0.000108 0.035992
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 4.591e-04 6.108e-05 7.517 1.01e-13 ***
L(ehatsq) 4.227e-01 2.453e-02 17.234 < 2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 0.00214 on 1365 degrees of freedom
Multiple R-squared: 0.1787, Adjusted R-squared: 0.1781
F-statistic: 297 on 1 and 1365 DF, p-value: < 2.2e-16
| Dependent variable: | |
| ehatsq | |
| Constant | 0.0005*** |
| (0.0001) | |
| L(ehatsq) | 0.423*** |
| (0.025) | |
| Observations | 1,367 |
| R2 | 0.179 |
| Adjusted R2 | 0.178 |
| Note: | p<0.1; p<0.05; p<0.01 |
library(broom)
T <- nobs(VALE.media)
q <- length(coef(VALE.ARCH))-1
Rsq <- glance(VALE.ARCH)[[1]]
LM <- (T-q)*Rsq
alpha <- 0.05
quiquadr <- qchisq(1-alpha, q)O valor da estatística LM é de 244.3 que deve ser comparado ao valor crítico do qui-quadrado com \(\alpha = 0,05\) e \(q=\) 1 grau de liberdade; este valor é \(\chi^{2}_{(0,95,\,\, 1)}\) = 3.84; isso indica que a hipótese nula é rejeitada, concluindo que a série tem efeitos ARCH.
A mesma conclusão pode ser alcançada se, em vez do procedimento passo a passo, usarmos um dos recursos de teste ARCH do R, a função ArchTest() no pacote FinTS.
library(FinTS)
VALEArchTest <- ArchTest(dlnVALEclose, lags=1, demean=TRUE)
VALEArchTest
ARCH LM-test; Null hypothesis: no ARCH effects
data: dlnVALEclose
Chi-squared = 244.3, df = 1, p-value < 2.2e-16A função garch() no pacote tseries torna-se um modelo ARCH quando usada com o order = argumento igual a c(0,1). Essa função pode ser usada para estimar e plotar a variância \(h_{t}\) definida na Equação 3, conforme mostrado no código a seguir e na Figura abaixo.
library(tseries)
rTS<-rTS%>%
na.omit() # Retiro os NAs da serie
VALE.arch <- garch(rTS,c(0,1))
***** ESTIMATION WITH ANALYTICAL GRADIENT *****
I INITIAL X(I) D(I)
1 7.555777e-04 1.000e+00
2 5.000000e-02 1.000e+00
IT NF F RELDF PRELDF RELDX STPPAR D*STEP NPRELDF
0 1 -4.247e+03
1 6 -4.252e+03 9.97e-04 2.18e-03 1.0e-03 9.3e+08 1.0e-04 1.01e+06
2 7 -4.252e+03 3.77e-05 4.69e-05 9.9e-04 2.0e+00 1.0e-04 3.39e+01
3 13 -4.271e+03 4.53e-03 7.62e-03 3.8e-01 2.0e+00 6.1e-02 3.38e+01
4 15 -4.273e+03 4.84e-04 1.16e-03 7.8e-02 1.1e+00 1.9e-02 1.85e-03
5 17 -4.279e+03 1.25e-03 8.55e-04 1.2e-01 0.0e+00 3.7e-02 8.55e-04
6 18 -4.281e+03 4.69e-04 4.06e-04 1.0e-01 1.8e-01 3.7e-02 4.15e-04
7 19 -4.281e+03 1.54e-04 1.17e-04 6.3e-02 0.0e+00 2.8e-02 1.17e-04
8 20 -4.281e+03 2.52e-05 2.17e-05 2.9e-02 0.0e+00 1.4e-02 2.17e-05
9 21 -4.281e+03 9.45e-07 8.79e-07 6.5e-03 0.0e+00 3.2e-03 8.79e-07
10 22 -4.281e+03 6.37e-09 6.26e-09 5.8e-04 0.0e+00 2.9e-04 6.26e-09
11 23 -4.281e+03 2.56e-12 2.53e-12 1.1e-05 0.0e+00 5.4e-06 2.53e-12
***** RELATIVE FUNCTION CONVERGENCE *****
FUNCTION -4.281430e+03 RELDX 1.083e-05
FUNC. EVALS 23 GRAD. EVALS 12
PRELDF 2.526e-12 NPRELDF 2.526e-12
I FINAL X(I) D(I) G(I)
1 5.627445e-04 1.000e+00 1.828e-01
2 2.475817e-01 1.000e+00 4.131e-05sbydarch <- summary(VALE.arch)
sbydarch
Call:
garch(x = rTS, order = c(0, 1))
Model:
GARCH(0,1)
Residuals:
Min 1Q Median 3Q Max
-4.68465 -0.59037 0.02321 0.59399 4.37563
Coefficient(s):
Estimate Std. Error t value Pr(>|t|)
a0 5.627e-04 2.198e-05 25.6 <2e-16 ***
a1 2.476e-01 2.293e-02 10.8 <2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Diagnostic Tests:
Jarque Bera Test
data: Residuals
X-squared = 134.77, df = 2, p-value < 2.2e-16
Box-Ljung test
data: Squared.Residuals
X-squared = 0.024545, df = 1, p-value = 0.8755hhat <- ts(2*VALE.arch$fitted.values[-1,1]^2)
plot.ts(hhat)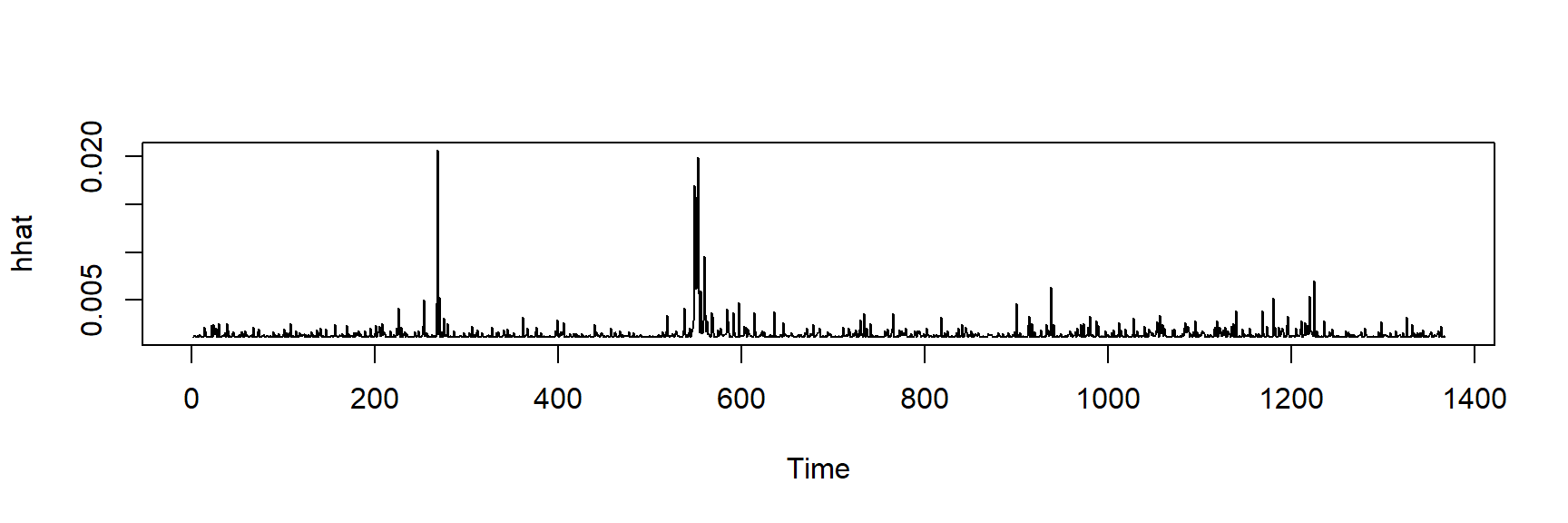
O modelo GARCH
library(rugarch)
garchSpec <- ugarchspec(
variance.model=list(model="sGARCH",
garchOrder=c(1,1)),
mean.model=list(armaOrder=c(0,0)),
distribution.model="std")
garchFit <- ugarchfit(spec=garchSpec, data=rTS)
coef(garchFit) mu omega alpha1 beta1 shape
5.098919e-04 8.181086e-05 7.732878e-02 8.075895e-01 6.563801e+00 rhat <- garchFit@fit$fitted.values
plot.ts(rhat)
hhat <- ts(garchFit@fit$sigma^2)
plot.ts(hhat)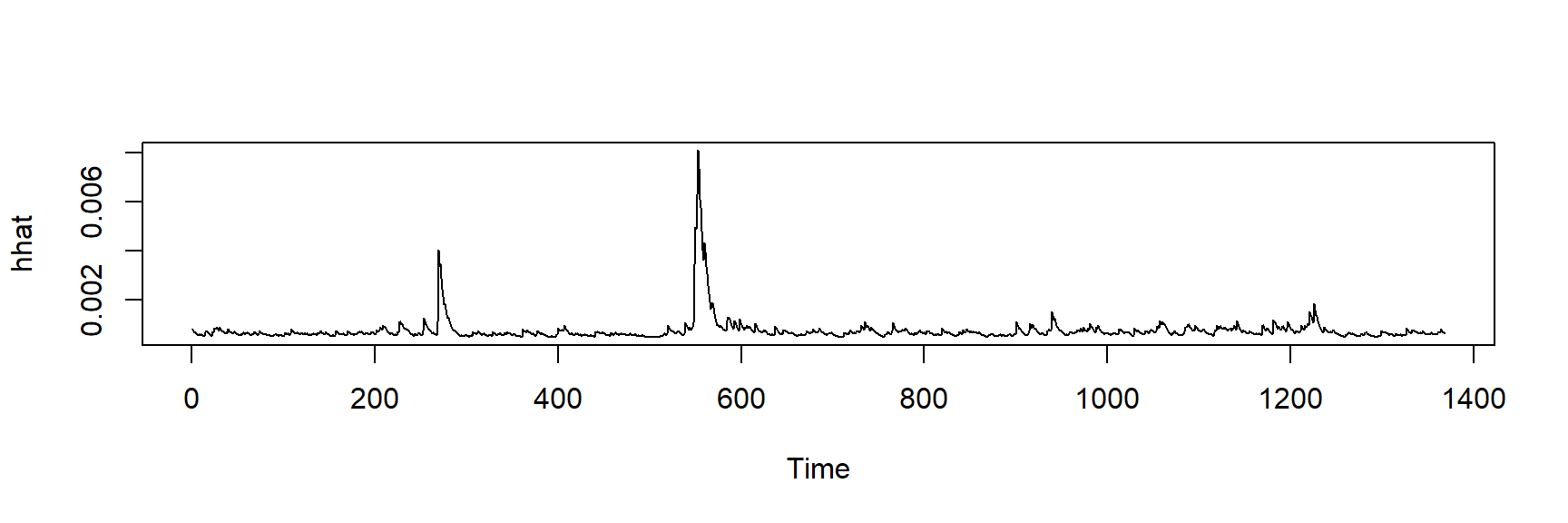
# tGARCH
garchMod <- ugarchspec(variance.model=list(model="fGARCH",
garchOrder=c(1,1),
submodel="TGARCH"),
mean.model=list(armaOrder=c(0,0)),
distribution.model="std")
garchFit <- ugarchfit(spec=garchMod, data=rTS)
coef(garchFit) mu omega alpha1 beta1 eta11 shape
0.0001448778 0.0024951666 0.0829903566 0.8426163519 0.6037352495 6.7955576554 rhat <- garchFit@fit$fitted.values
plot.ts(rhat)
hhat <- ts(garchFit@fit$sigma^2)
plot.ts(hhat)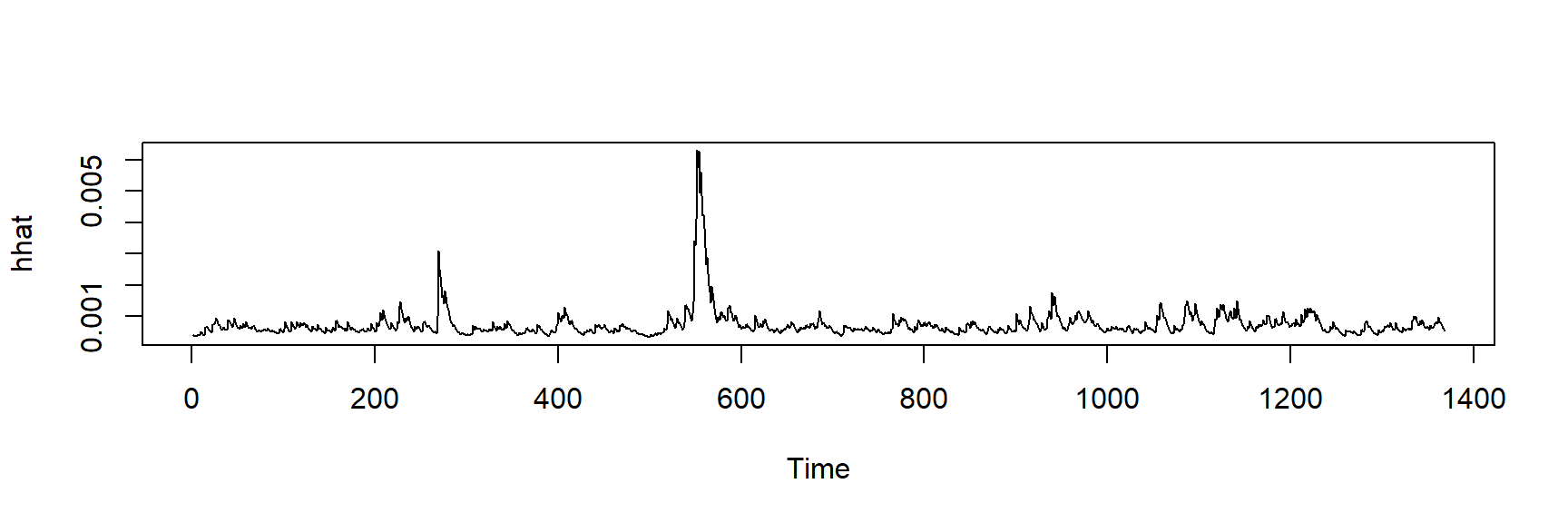
# GARCH-in-mean
garchMod <- ugarchspec(
variance.model=list(model="fGARCH",
garchOrder=c(1,1),
submodel="APARCH"),
mean.model=list(armaOrder=c(0,0),
include.mean=TRUE,
archm=TRUE,
archpow=2
),
distribution.model="std"
)
garchFit <- ugarchfit(spec=garchMod, data=rTS)
coef(garchFit) mu archm omega alpha1 beta1 eta11
-0.001572400 2.693463496 0.001299905 0.083006027 0.814291951 0.578064680
lambda shape
1.237831744 6.936388133 rhat <- garchFit@fit$fitted.values
plot.ts(rhat)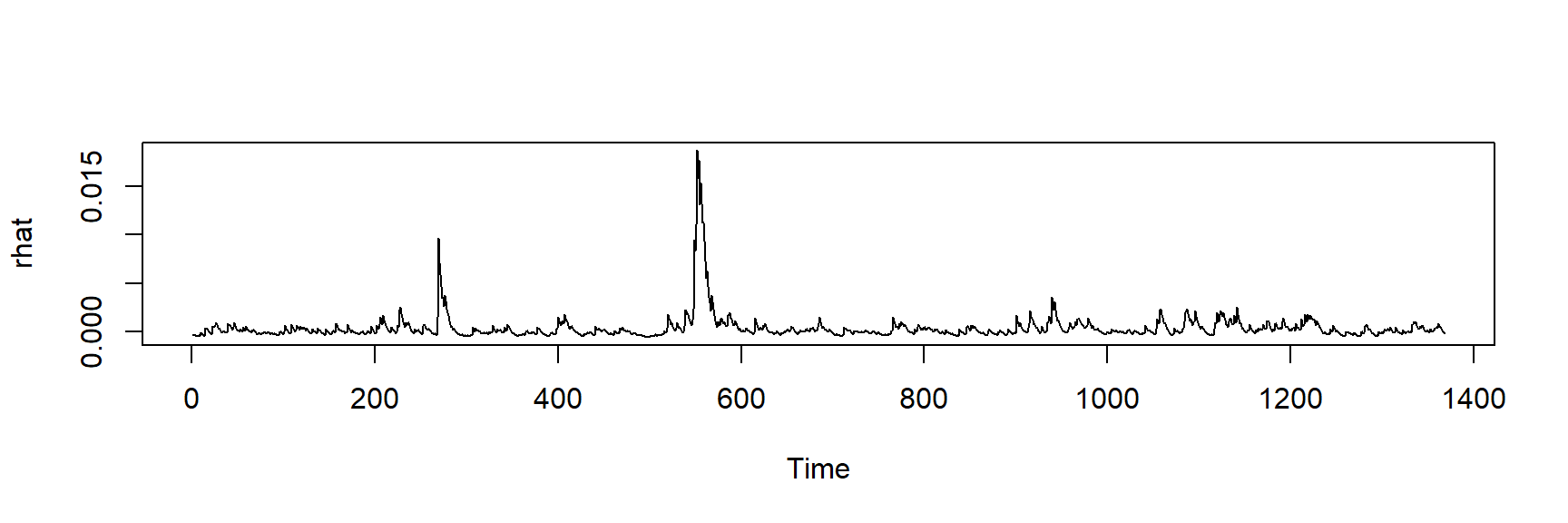
hhat <- ts(garchFit@fit$sigma^2)
plot.ts(hhat)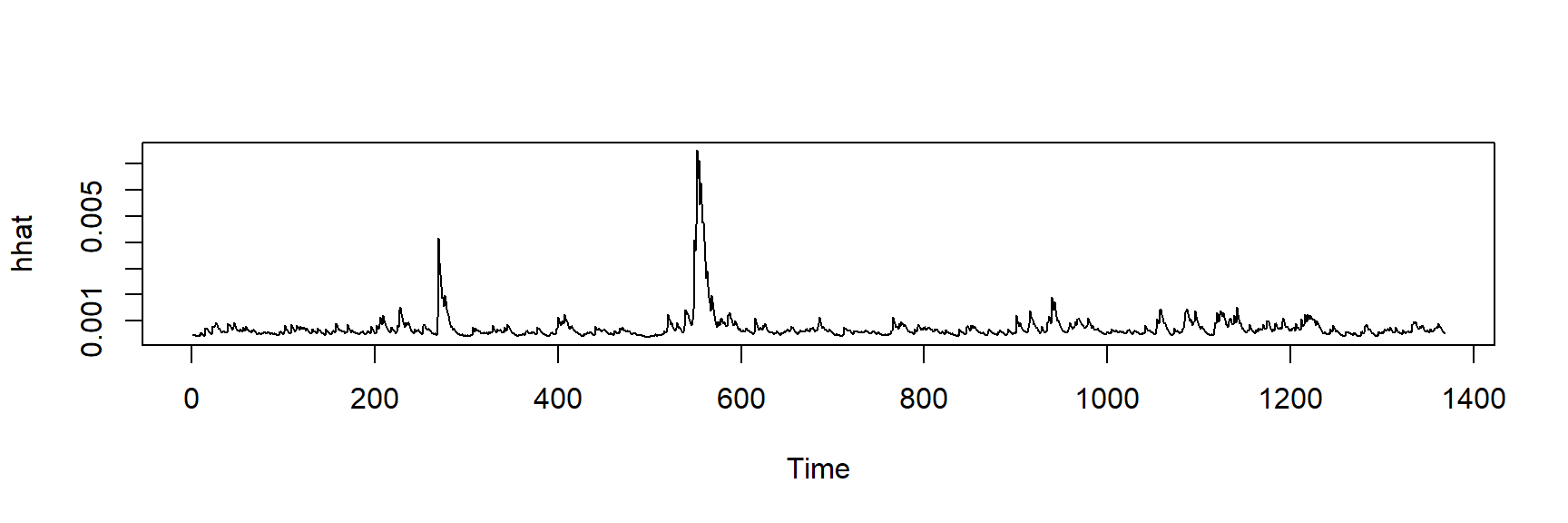
As Figuras anteriores mostram algumas versões do modelo GARCH. As previsões podem ser obtidas usando a função ugarchboot() do pacote ugarch.
Outro script para a News Impact Curve
library(PerformanceAnalytics)
library(quantmod)
library(rugarch)
library(car)
library(FinTS)options(digits=4)
VALEfechamento<-ativo2$VALE.Close
# Calcula o log retorno dos precos de fechamento da VALE
VALE.ret = CalculateReturns(VALEfechamento, method="log")
VALE.ret[1:10,] # printa as primeiras dez linhas VALE.Close
2018-01-02 NA
2018-01-03 0.006245
2018-01-04 -0.001558
2018-01-05 0.020062
2018-01-08 0.017418
2018-01-09 -0.006024
2018-01-10 -0.006821
2018-01-11 0.022557
2018-01-12 0.005930
2018-01-16 -0.026968VALE.ret=VALE.ret[-1,] # Removo o primeiro NA
VALE.ret[1:10,] # printa as primeiras dez linhas VALE.Close
2018-01-03 0.006245
2018-01-04 -0.001558
2018-01-05 0.020062
2018-01-08 0.017418
2018-01-09 -0.006024
2018-01-10 -0.006821
2018-01-11 0.022557
2018-01-12 0.005930
2018-01-16 -0.026968
2018-01-17 0.013575GARCH Assimétrico
Agora vamos definir o GARCH assimétrico
garch11.spec = ugarchspec(variance.model = list(garchOrder=c(1,1)),
mean.model = list(armaOrder=c(0,0)))
VALE.garch11.fit = ugarchfit(spec=garch11.spec, data=VALE.ret,
solver.control=list(trace = 1))
Iter: 1 fn: -3045.0634 Pars: 0.0005617 0.0001112 0.1226330 0.7247662
Iter: 2 fn: -3045.0634 Pars: 0.0005616 0.0001112 0.1226429 0.7247721
solnp--> Completed in 2 iterationsVALE.garch11.fit
*---------------------------------*
* GARCH Model Fit *
*---------------------------------*
Conditional Variance Dynamics
-----------------------------------
GARCH Model : sGARCH(1,1)
Mean Model : ARFIMA(0,0,0)
Distribution : norm
Optimal Parameters
------------------------------------
Estimate Std. Error t value Pr(>|t|)
mu 0.000562 0.000676 0.83051 0.406253
omega 0.000111 0.000034 3.27136 0.001070
alpha1 0.122643 0.024780 4.94933 0.000001
beta1 0.724772 0.063199 11.46801 0.000000
Robust Standard Errors:
Estimate Std. Error t value Pr(>|t|)
mu 0.000562 0.000667 0.84239 0.399568
omega 0.000111 0.000060 1.84512 0.065019
alpha1 0.122643 0.059972 2.04499 0.040856
beta1 0.724772 0.124365 5.82780 0.000000
LogLikelihood : 3045
Information Criteria
------------------------------------
Akaike -4.4460
Bayes -4.4307
Shibata -4.4460
Hannan-Quinn -4.4403
Weighted Ljung-Box Test on Standardized Residuals
------------------------------------
statistic p-value
Lag[1] 0.01975 0.8882
Lag[2*(p+q)+(p+q)-1][2] 0.23233 0.8346
Lag[4*(p+q)+(p+q)-1][5] 1.50811 0.7379
d.o.f=0
H0 : No serial correlation
Weighted Ljung-Box Test on Standardized Squared Residuals
------------------------------------
statistic p-value
Lag[1] 6.808 0.009077
Lag[2*(p+q)+(p+q)-1][5] 7.745 0.034073
Lag[4*(p+q)+(p+q)-1][9] 9.341 0.069102
d.o.f=2
Weighted ARCH LM Tests
------------------------------------
Statistic Shape Scale P-Value
ARCH Lag[3] 0.090 0.500 2.000 0.7642
ARCH Lag[5] 1.151 1.440 1.667 0.6888
ARCH Lag[7] 1.996 2.315 1.543 0.7181
Nyblom stability test
------------------------------------
Joint Statistic: 0.6963
Individual Statistics:
mu 0.05934
omega 0.17910
alpha1 0.07439
beta1 0.20613
Asymptotic Critical Values (10% 5% 1%)
Joint Statistic: 1.07 1.24 1.6
Individual Statistic: 0.35 0.47 0.75
Sign Bias Test
------------------------------------
t-value prob sig
Sign Bias 1.1389 0.254925
Negative Sign Bias 3.1327 0.001769 ***
Positive Sign Bias 0.8256 0.409155
Joint Effect 12.0285 0.007286 ***
Adjusted Pearson Goodness-of-Fit Test:
------------------------------------
group statistic p-value(g-1)
1 20 38.67 0.004877
2 30 43.18 0.043775
3 40 57.32 0.029368
4 50 72.64 0.015750
Elapsed time : 0.1835 plot(residuals(VALE.garch11.fit))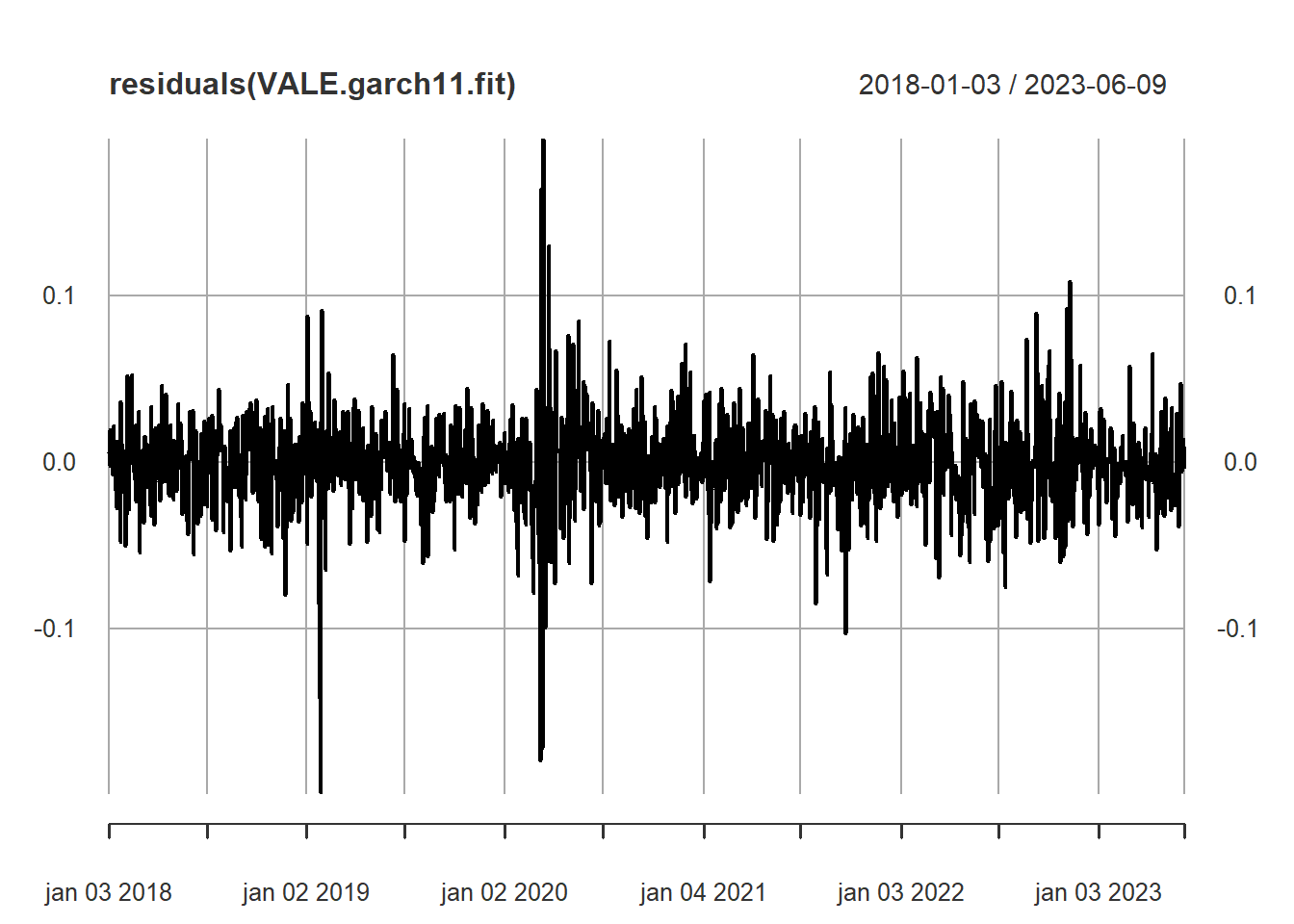
Rodaremos o teste de Engle & NG. para o viés de sinal:
O teste de viés de sinal considera a variável \(S_{t-1}^{-}\), uma variável fictícia que assume o valor um quando \(\epsilon_{t-1}\) é negativo e zero, caso contrário. Este teste examina o impacto de choques de retorno positivo e negativo sobre a volatilidade não prevista pelo modelo em consideração. O teste de viés de tamanho negativo utiliza a variável \(S_{t-1}^{-}\) \(\epsilon_{t-1}\). Ele se concentra nos diferentes efeitos que grandes e pequenos choques de retorno negativo têm sobre a volatilidade, o que não é previsto pelo modelo de volatilidade. O teste de viés de tamanho positivo utiliza a variável \(S_{t-1}^{+}\) \(\epsilon_{t-1}\) onde \(S_{t-1}^{+}\) é definido como 1 menos \(S_{t-1}\). Ele se concentra nos diferentes impactos que grandes e pequenos choques de retorno positivo podem ter sobre a volatilidade, que não são explicados pelo modelo de volatilidade.
signbias(VALE.garch11.fit) t-value prob sig
Sign Bias 1.1389 0.254925
Negative Sign Bias 3.1327 0.001769 ***
Positive Sign Bias 0.8256 0.409155
Joint Effect 12.0285 0.007286 ***Como apontado acima, o efeito de viés de negativo foi o que apresentou significância.
Modelo EGARCH de Nelson
egarch11.spec = ugarchspec(variance.model=list(model="eGARCH",
garchOrder=c(1,1)),
mean.model=list(armaOrder=c(0,0)))
VALE.egarch11.fit = ugarchfit(egarch11.spec, VALE.ret)
VALE.egarch11.fit
*---------------------------------*
* GARCH Model Fit *
*---------------------------------*
Conditional Variance Dynamics
-----------------------------------
GARCH Model : eGARCH(1,1)
Mean Model : ARFIMA(0,0,0)
Distribution : norm
Optimal Parameters
------------------------------------
Estimate Std. Error t value Pr(>|t|)
mu -0.000209 0.000671 -0.31211 0.754954
omega -0.944470 0.262859 -3.59307 0.000327
alpha1 -0.132769 0.027603 -4.80997 0.000002
beta1 0.869359 0.036082 24.09385 0.000000
gamma1 0.213962 0.035379 6.04777 0.000000
Robust Standard Errors:
Estimate Std. Error t value Pr(>|t|)
mu -0.000209 0.000705 -0.2969 0.766544
omega -0.944470 0.573335 -1.6473 0.099491
alpha1 -0.132769 0.059557 -2.2293 0.025796
beta1 0.869359 0.079300 10.9629 0.000000
gamma1 0.213962 0.076979 2.7795 0.005445
LogLikelihood : 3054
Information Criteria
------------------------------------
Akaike -4.4573
Bayes -4.4382
Shibata -4.4573
Hannan-Quinn -4.4501
Weighted Ljung-Box Test on Standardized Residuals
------------------------------------
statistic p-value
Lag[1] 3.039e-06 0.9986
Lag[2*(p+q)+(p+q)-1][2] 1.742e-01 0.8703
Lag[4*(p+q)+(p+q)-1][5] 1.226e+00 0.8068
d.o.f=0
H0 : No serial correlation
Weighted Ljung-Box Test on Standardized Squared Residuals
------------------------------------
statistic p-value
Lag[1] 6.503 0.01077
Lag[2*(p+q)+(p+q)-1][5] 6.939 0.05367
Lag[4*(p+q)+(p+q)-1][9] 8.567 0.09955
d.o.f=2
Weighted ARCH LM Tests
------------------------------------
Statistic Shape Scale P-Value
ARCH Lag[3] 0.09855 0.500 2.000 0.7536
ARCH Lag[5] 0.49815 1.440 1.667 0.8842
ARCH Lag[7] 1.95431 2.315 1.543 0.7270
Nyblom stability test
------------------------------------
Joint Statistic: 0.8103
Individual Statistics:
mu 0.1289
omega 0.1249
alpha1 0.1322
beta1 0.1188
gamma1 0.2905
Asymptotic Critical Values (10% 5% 1%)
Joint Statistic: 1.28 1.47 1.88
Individual Statistic: 0.35 0.47 0.75
Sign Bias Test
------------------------------------
t-value prob sig
Sign Bias 1.19739 0.23136
Negative Sign Bias 2.22994 0.02591 **
Positive Sign Bias 0.05336 0.95745
Joint Effect 4.99000 0.17253
Adjusted Pearson Goodness-of-Fit Test:
------------------------------------
group statistic p-value(g-1)
1 20 33.23 0.02262
2 30 44.98 0.02954
3 40 50.36 0.10506
4 50 69.35 0.02935
Elapsed time : 0.1737 plot(residuals(VALE.egarch11.fit))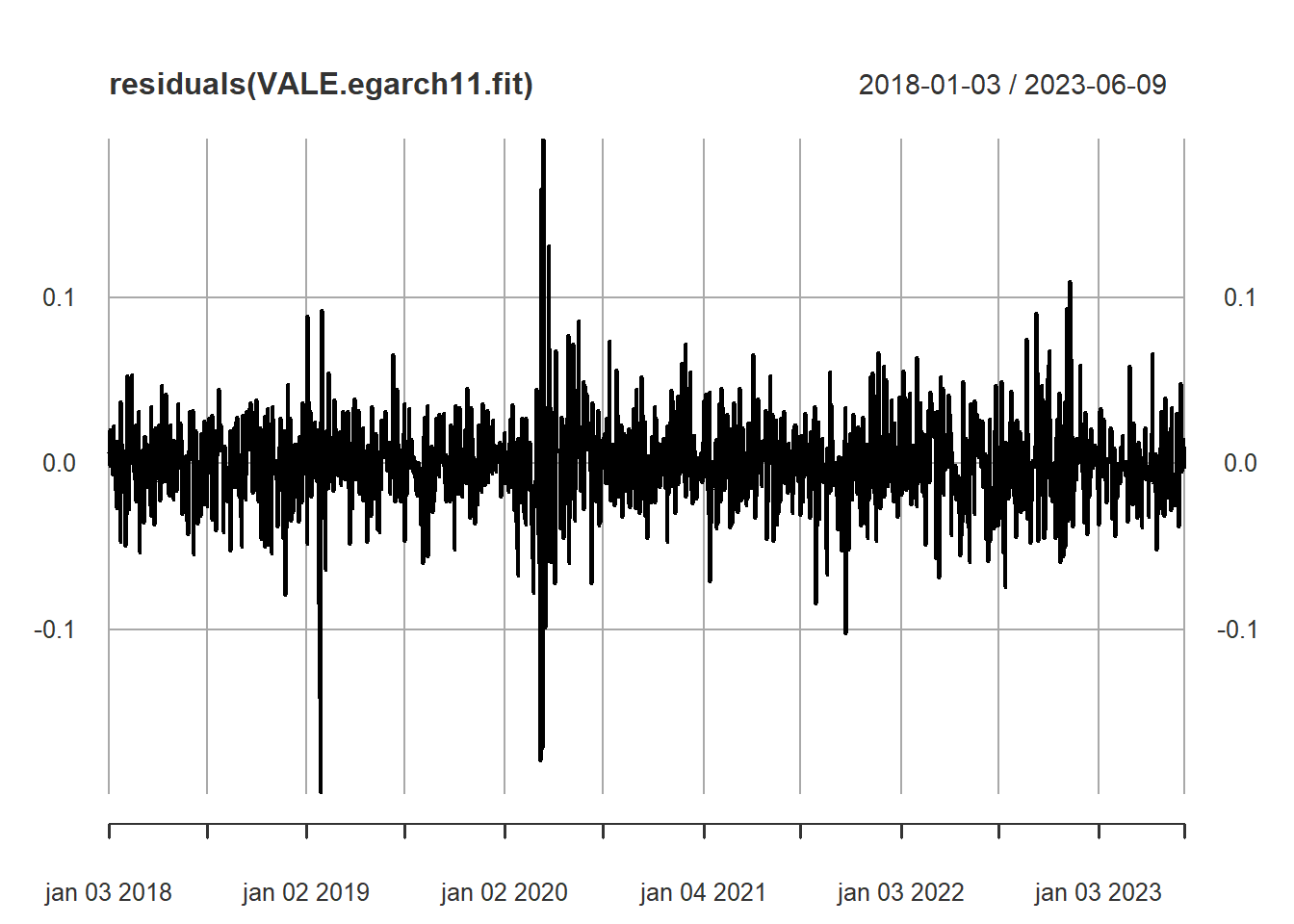
Modelo GJR GARCH
gjrgarch11.spec = ugarchspec(variance.model=list(model="gjrGARCH",
garchOrder=c(1,1)),
mean.model=list(armaOrder=c(0,0)))
VALE.gjrgarch11.fit = ugarchfit(gjrgarch11.spec, VALE.ret)
VALE.gjrgarch11.fit
*---------------------------------*
* GARCH Model Fit *
*---------------------------------*
Conditional Variance Dynamics
-----------------------------------
GARCH Model : gjrGARCH(1,1)
Mean Model : ARFIMA(0,0,0)
Distribution : norm
Optimal Parameters
------------------------------------
Estimate Std. Error t value Pr(>|t|)
mu 0.000047 0.000677 0.068915 0.945057
omega 0.000128 0.000032 4.002958 0.000063
alpha1 0.013622 0.021287 0.639913 0.522229
beta1 0.711888 0.056802 12.532797 0.000000
gamma1 0.192620 0.046233 4.166272 0.000031
Robust Standard Errors:
Estimate Std. Error t value Pr(>|t|)
mu 0.000047 0.000702 0.06647 0.947004
omega 0.000128 0.000056 2.29011 0.022015
alpha1 0.013622 0.021603 0.63055 0.528333
beta1 0.711888 0.102368 6.95423 0.000000
gamma1 0.192620 0.094309 2.04243 0.041109
LogLikelihood : 3057
Information Criteria
------------------------------------
Akaike -4.4618
Bayes -4.4427
Shibata -4.4618
Hannan-Quinn -4.4547
Weighted Ljung-Box Test on Standardized Residuals
------------------------------------
statistic p-value
Lag[1] 0.007495 0.9310
Lag[2*(p+q)+(p+q)-1][2] 0.098210 0.9209
Lag[4*(p+q)+(p+q)-1][5] 1.211921 0.8101
d.o.f=0
H0 : No serial correlation
Weighted Ljung-Box Test on Standardized Squared Residuals
------------------------------------
statistic p-value
Lag[1] 3.171 0.07496
Lag[2*(p+q)+(p+q)-1][5] 4.090 0.24311
Lag[4*(p+q)+(p+q)-1][9] 6.367 0.25817
d.o.f=2
Weighted ARCH LM Tests
------------------------------------
Statistic Shape Scale P-Value
ARCH Lag[3] 0.2943 0.500 2.000 0.5875
ARCH Lag[5] 1.1262 1.440 1.667 0.6959
ARCH Lag[7] 3.0940 2.315 1.543 0.4960
Nyblom stability test
------------------------------------
Joint Statistic: 0.9498
Individual Statistics:
mu 0.1273
omega 0.2589
alpha1 0.1830
beta1 0.3513
gamma1 0.1092
Asymptotic Critical Values (10% 5% 1%)
Joint Statistic: 1.28 1.47 1.88
Individual Statistic: 0.35 0.47 0.75
Sign Bias Test
------------------------------------
t-value prob sig
Sign Bias 0.6108 0.5414
Negative Sign Bias 1.5094 0.1314
Positive Sign Bias 0.2721 0.7856
Joint Effect 2.3523 0.5026
Adjusted Pearson Goodness-of-Fit Test:
------------------------------------
group statistic p-value(g-1)
1 20 33.81 0.01933
2 30 39.41 0.09401
3 40 49.37 0.12351
4 50 64.75 0.06522
Elapsed time : 0.3573 plot(residuals(VALE.gjrgarch11.fit))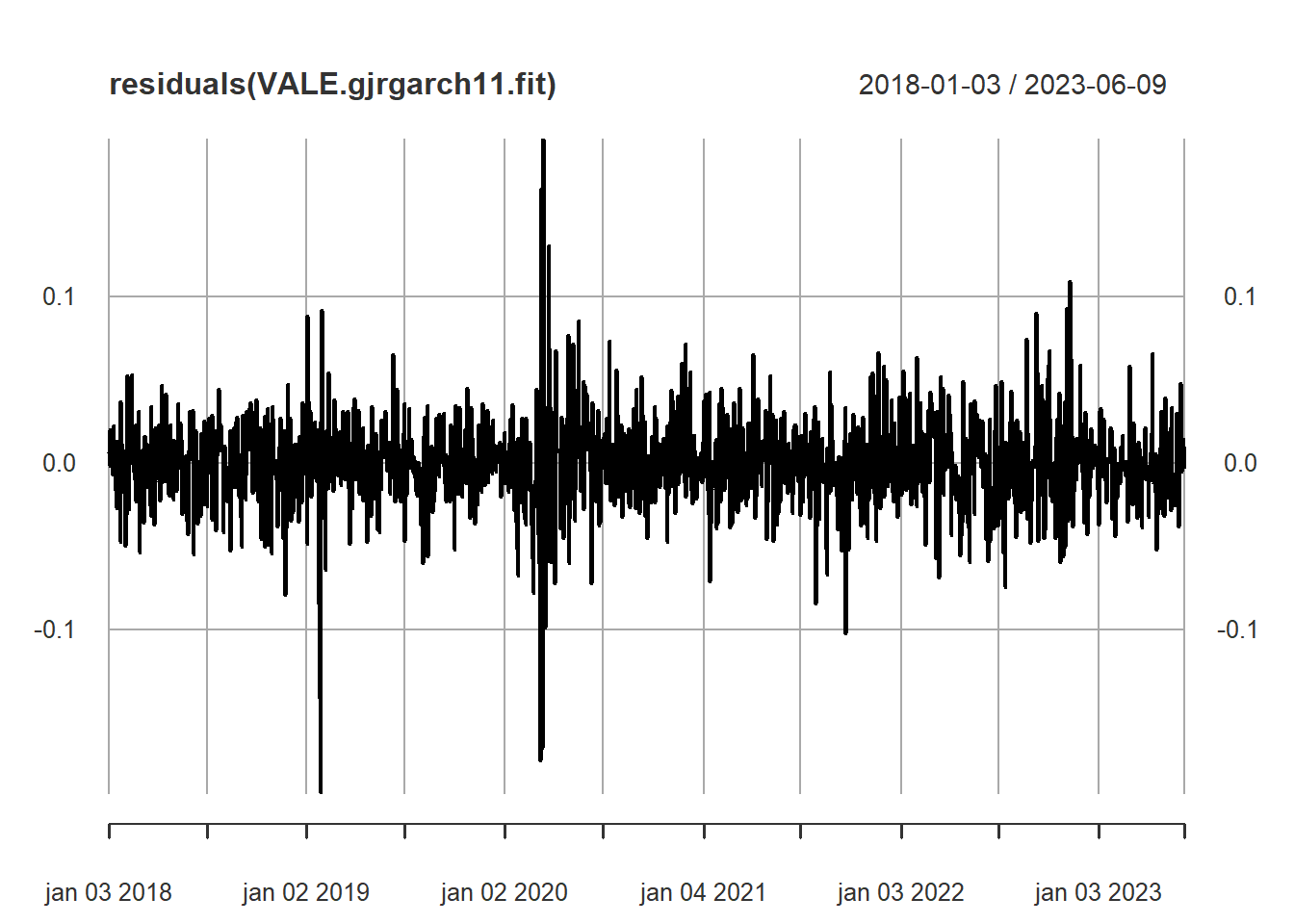
Modelo APARCH
aparch11.1.spec = ugarchspec(variance.model=list(model="apARCH",
garchOrder=c(1,1)),
mean.model=list(armaOrder=c(0,0)),
fixed.pars=list(delta=1))
VALE.aparch11.1.fit = ugarchfit(aparch11.1.spec, VALE.ret)
VALE.aparch11.1.fit
*---------------------------------*
* GARCH Model Fit *
*---------------------------------*
Conditional Variance Dynamics
-----------------------------------
GARCH Model : apARCH(1,1)
Mean Model : ARFIMA(0,0,0)
Distribution : norm
Optimal Parameters
------------------------------------
Estimate Std. Error t value Pr(>|t|)
mu -0.000214 0.000673 -0.31766 0.750739
omega 0.004021 0.001095 3.67160 0.000241
alpha1 0.116203 0.020798 5.58731 0.000000
beta1 0.760789 0.051128 14.87997 0.000000
gamma1 0.653656 0.150914 4.33131 0.000015
delta 1.000000 NA NA NA
Robust Standard Errors:
Estimate Std. Error t value Pr(>|t|)
mu -0.000214 0.000703 -0.30417 0.761000
omega 0.004021 0.002296 1.75105 0.079937
alpha1 0.116203 0.042770 2.71689 0.006590
beta1 0.760789 0.112343 6.77204 0.000000
gamma1 0.653656 0.166548 3.92472 0.000087
delta 1.000000 NA NA NA
LogLikelihood : 3054
Information Criteria
------------------------------------
Akaike -4.4573
Bayes -4.4382
Shibata -4.4573
Hannan-Quinn -4.4501
Weighted Ljung-Box Test on Standardized Residuals
------------------------------------
statistic p-value
Lag[1] 0.001546 0.9686
Lag[2*(p+q)+(p+q)-1][2] 0.251705 0.8232
Lag[4*(p+q)+(p+q)-1][5] 1.405315 0.7632
d.o.f=0
H0 : No serial correlation
Weighted Ljung-Box Test on Standardized Squared Residuals
------------------------------------
statistic p-value
Lag[1] 9.593 0.001953
Lag[2*(p+q)+(p+q)-1][5] 9.929 0.009601
Lag[4*(p+q)+(p+q)-1][9] 11.523 0.023244
d.o.f=2
Weighted ARCH LM Tests
------------------------------------
Statistic Shape Scale P-Value
ARCH Lag[3] 0.06548 0.500 2.000 0.7980
ARCH Lag[5] 0.59218 1.440 1.667 0.8561
ARCH Lag[7] 1.97026 2.315 1.543 0.7236
Nyblom stability test
------------------------------------
Joint Statistic: 0.7544
Individual Statistics:
mu 0.13466
omega 0.08956
alpha1 0.09893
beta1 0.10852
gamma1 0.12171
Asymptotic Critical Values (10% 5% 1%)
Joint Statistic: 1.28 1.47 1.88
Individual Statistic: 0.35 0.47 0.75
Sign Bias Test
------------------------------------
t-value prob sig
Sign Bias 1.461350 0.14415
Negative Sign Bias 2.570240 0.01027 **
Positive Sign Bias 0.002776 0.99779
Joint Effect 6.651720 0.08387 *
Adjusted Pearson Goodness-of-Fit Test:
------------------------------------
group statistic p-value(g-1)
1 20 35.42 0.012422
2 30 43.97 0.036913
3 40 50.54 0.102050
4 50 75.06 0.009726
Elapsed time : 0.4172 plot(residuals(VALE.aparch11.1.fit))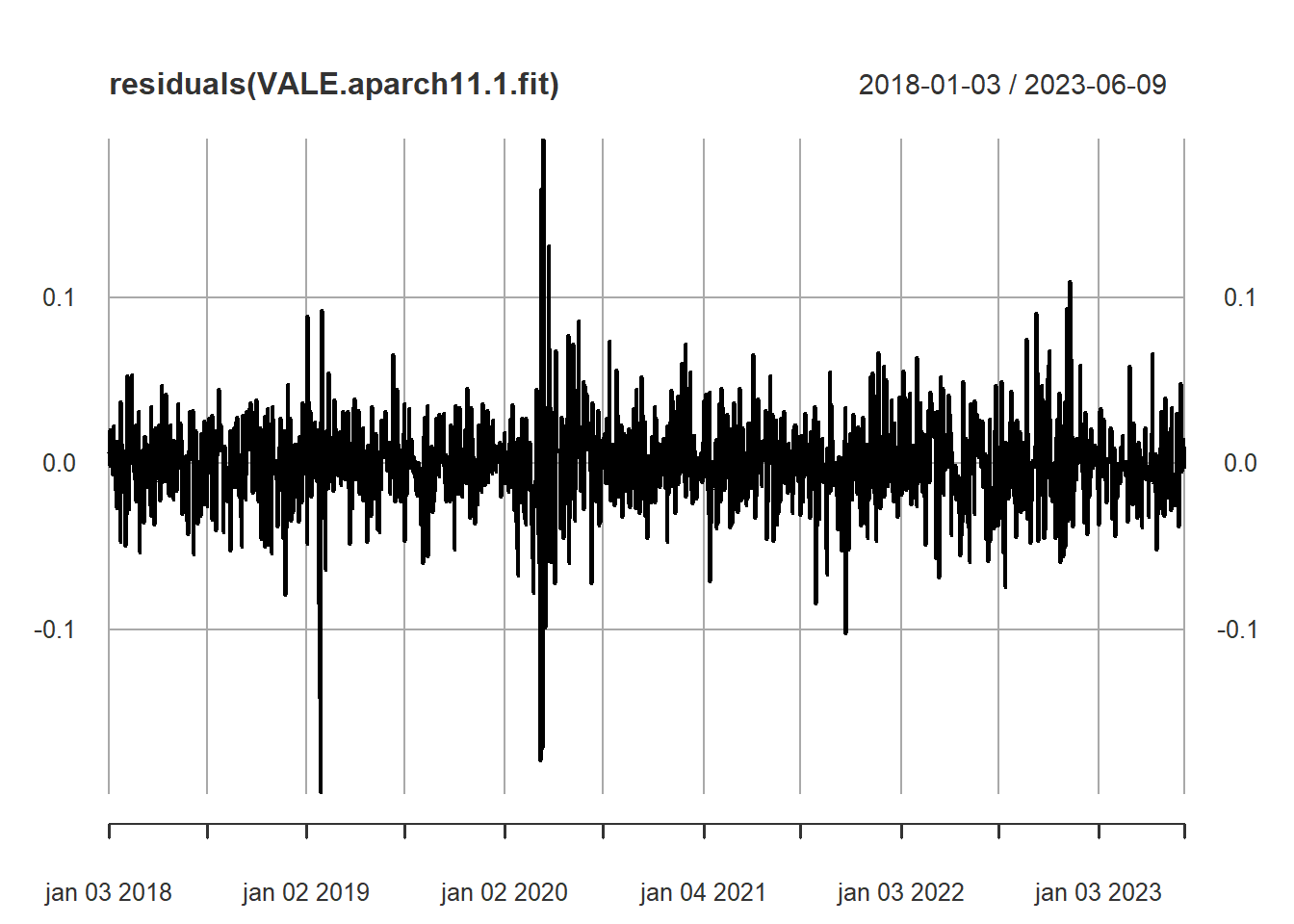
Comparando as News Impact Curves
nic.garch11 = newsimpact(VALE.garch11.fit)
nic.egarch11 = newsimpact(VALE.egarch11.fit)
nic.gjrgarch11 = newsimpact(VALE.gjrgarch11.fit)
nic.aparch11.1 = newsimpact(VALE.aparch11.1.fit)Via critério de informação, comparamos:
model.list = list(garch11 = VALE.garch11.fit,
egarch11 = VALE.egarch11.fit,
gjrgarch11 = VALE.gjrgarch11.fit,
aparch11.1 = VALE.aparch11.1.fit)
info.mat = sapply(model.list, infocriteria)
rownames(info.mat) = rownames(infocriteria(VALE.garch11.fit))
info.mat garch11 egarch11 gjrgarch11 aparch11.1
Akaike -4.446 -4.457 -4.462 -4.457
Bayes -4.431 -4.438 -4.443 -4.438
Shibata -4.446 -4.457 -4.462 -4.457
Hannan-Quinn -4.440 -4.450 -4.455 -4.450Mostra a News Impact Curve estimada GARCH(1,1) e EGARCH(1,1)
par(mfrow=c(2,2))
plot(nic.garch11$zx, type="l", lwd=2, col="blue", main="GARCH(1,1)",
nic.garch11$zy, ylab=nic.garch11$yexpr, xlab=nic.garch11$xexpr)
plot(nic.egarch11$zx, type="l", lwd=2, col="blue", main="EGARCH(1,1)",
nic.egarch11$zy, ylab=nic.egarch11$yexpr, xlab=nic.egarch11$xexpr)
plot(nic.gjrgarch11$zx, type="l", lwd=2, col="blue", main="TGARCH(1,1)",
nic.gjrgarch11$zy, ylab=nic.gjrgarch11$yexpr, xlab=nic.gjrgarch11$xexpr)
plot(nic.aparch11.1$zx, type="l", lwd=2, col="blue", main="APARCH(1,1,1)",
nic.aparch11.1$zy, ylab=nic.aparch11.1$yexpr, xlab=nic.aparch11.1$xexpr)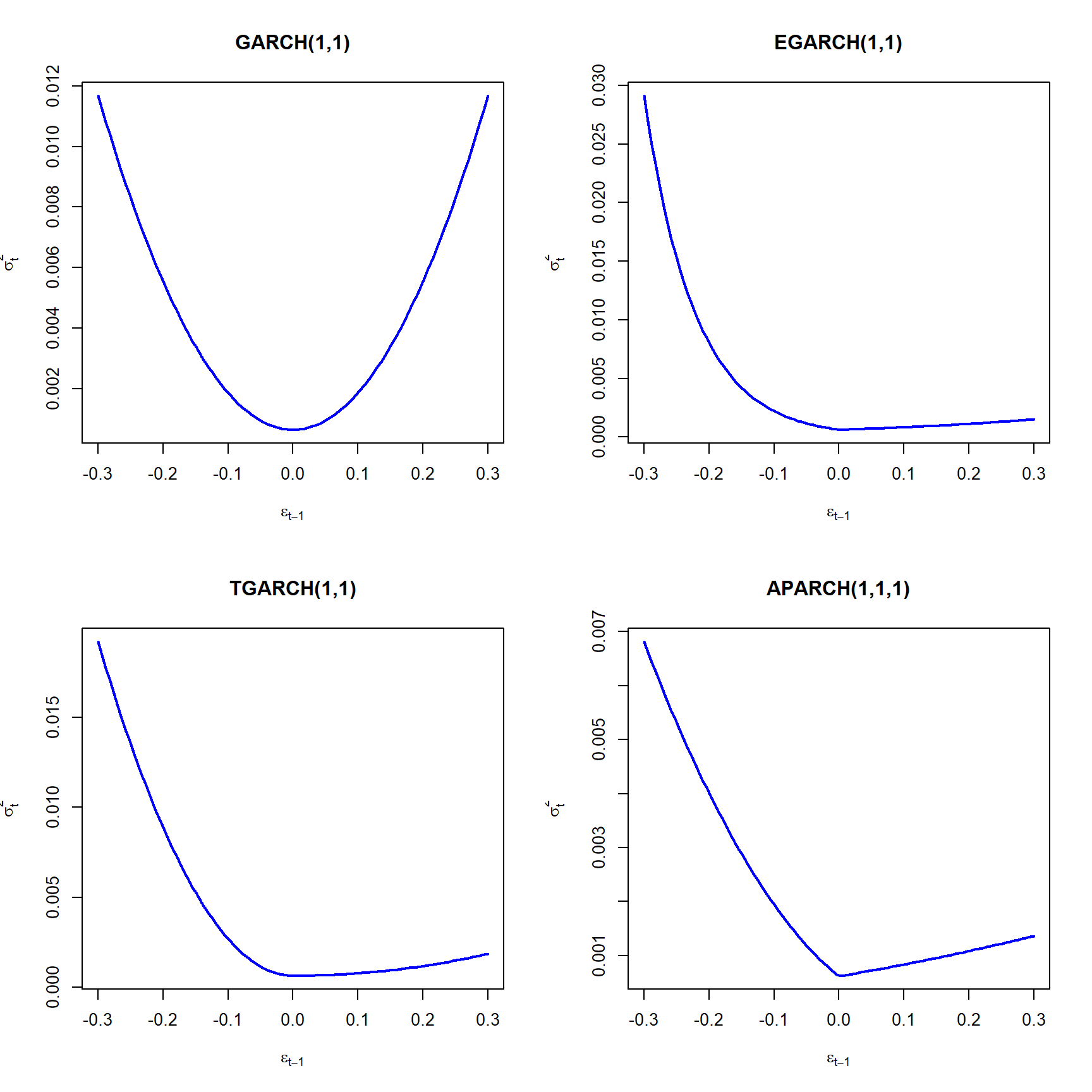
Modelo GARCH com erros não-normais
Vamos observar um modelo GARCH(1,1) normal, examinaremos os resíduos padronizados:
VALE.garch11.zt = residuals(VALE.garch11.fit)/sigma(VALE.garch11.fit)
VALE.garch11.zt = xts(VALE.garch11.zt, order.by=index(VALE.ret))
qqPlot(coredata(VALE.garch11.zt))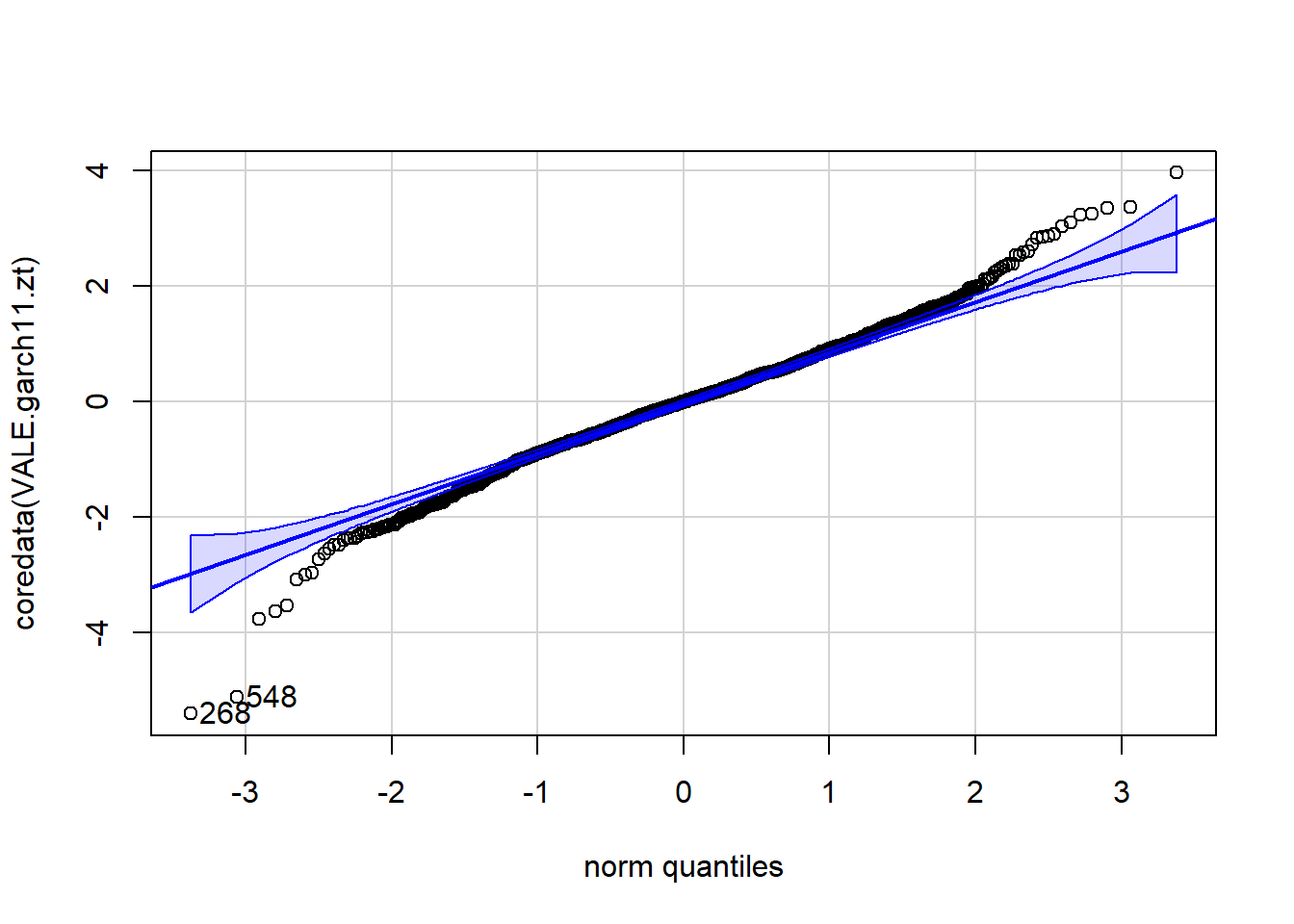
[1] 268 548plot(VALE.garch11.fit, which=8)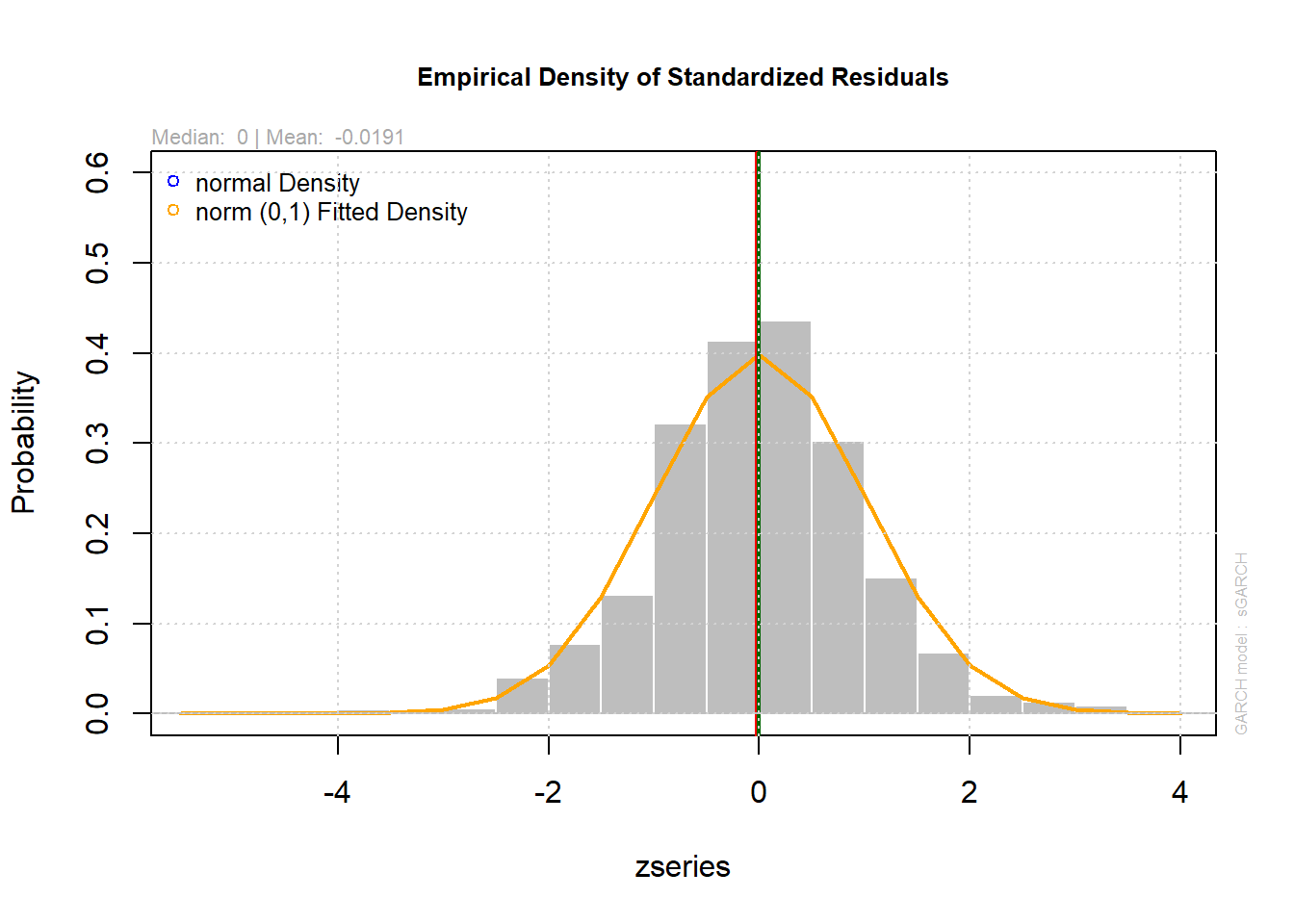
plot(VALE.garch11.fit, which=9)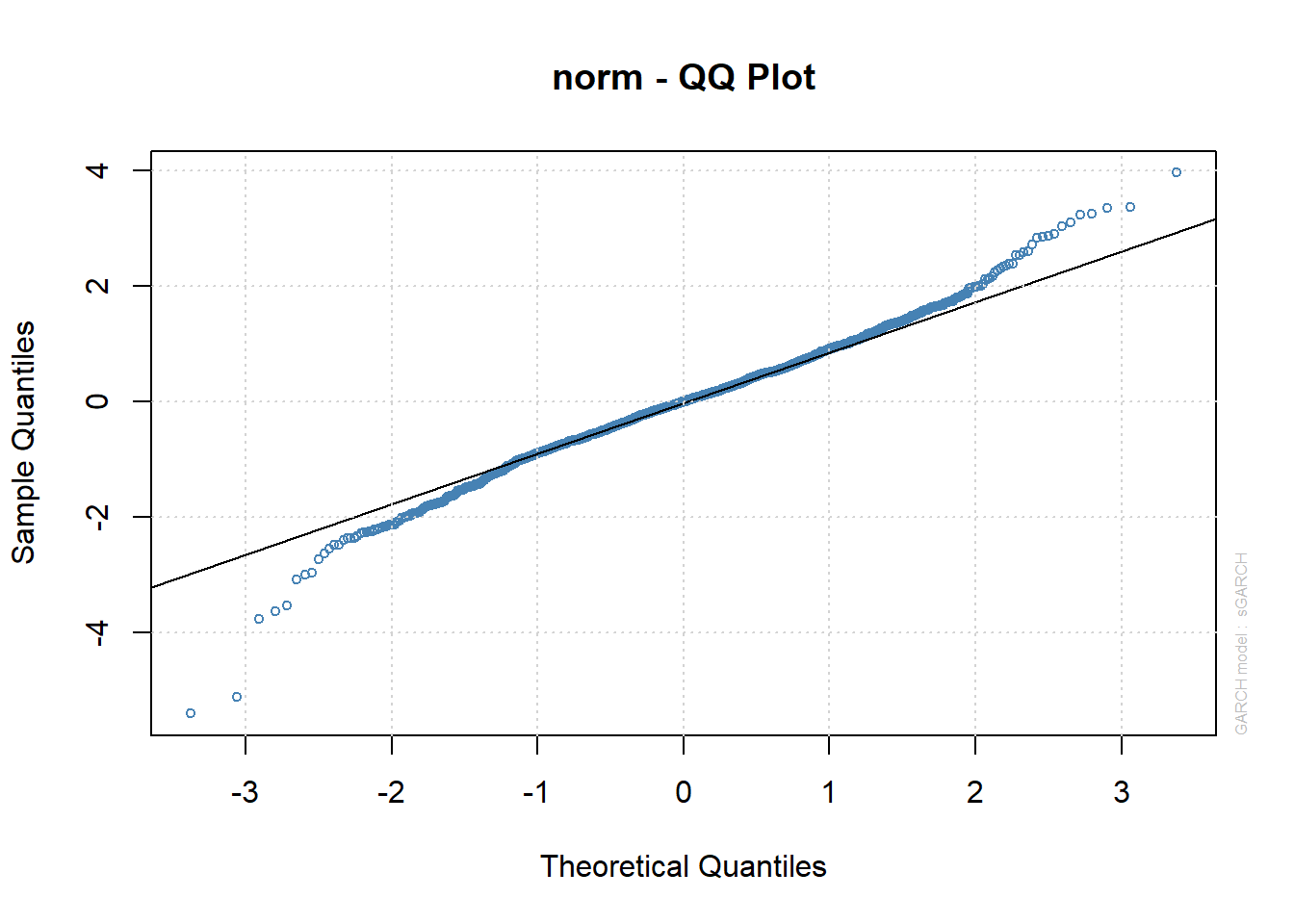
Rodaremos um modelo GARCH(1,1) com erros de Student \(t\):
garch11.t.spec = ugarchspec(variance.model = list(garchOrder=c(1,1)),
mean.model = list(armaOrder=c(0,0)),
distribution.model = "std")
VALE.garch11.t.fit = ugarchfit(spec=garch11.t.spec, data=VALE.ret)
VALE.garch11.t.fit
*---------------------------------*
* GARCH Model Fit *
*---------------------------------*
Conditional Variance Dynamics
-----------------------------------
GARCH Model : sGARCH(1,1)
Mean Model : ARFIMA(0,0,0)
Distribution : std
Optimal Parameters
------------------------------------
Estimate Std. Error t value Pr(>|t|)
mu 0.000510 0.000646 0.78967 0.429722
omega 0.000082 0.000032 2.52697 0.011505
alpha1 0.077329 0.023052 3.35461 0.000795
beta1 0.807590 0.060547 13.33824 0.000000
shape 6.563801 1.125766 5.83052 0.000000
Robust Standard Errors:
Estimate Std. Error t value Pr(>|t|)
mu 0.000510 0.000633 0.8060 0.420244
omega 0.000082 0.000035 2.3304 0.019786
alpha1 0.077329 0.037766 2.0476 0.040604
beta1 0.807590 0.072977 11.0664 0.000000
shape 6.563801 1.222901 5.3674 0.000000
LogLikelihood : 3075
Information Criteria
------------------------------------
Akaike -4.4887
Bayes -4.4696
Shibata -4.4887
Hannan-Quinn -4.4815
Weighted Ljung-Box Test on Standardized Residuals
------------------------------------
statistic p-value
Lag[1] 0.003734 0.9513
Lag[2*(p+q)+(p+q)-1][2] 0.281145 0.8063
Lag[4*(p+q)+(p+q)-1][5] 1.695005 0.6920
d.o.f=0
H0 : No serial correlation
Weighted Ljung-Box Test on Standardized Squared Residuals
------------------------------------
statistic p-value
Lag[1] 15.55 8.029e-05
Lag[2*(p+q)+(p+q)-1][5] 15.95 2.460e-04
Lag[4*(p+q)+(p+q)-1][9] 17.14 1.066e-03
d.o.f=2
Weighted ARCH LM Tests
------------------------------------
Statistic Shape Scale P-Value
ARCH Lag[3] 0.3973 0.500 2.000 0.5285
ARCH Lag[5] 0.6670 1.440 1.667 0.8335
ARCH Lag[7] 1.2800 2.315 1.543 0.8642
Nyblom stability test
------------------------------------
Joint Statistic: 0.8766
Individual Statistics:
mu 0.1006
omega 0.2864
alpha1 0.2121
beta1 0.3080
shape 0.3454
Asymptotic Critical Values (10% 5% 1%)
Joint Statistic: 1.28 1.47 1.88
Individual Statistic: 0.35 0.47 0.75
Sign Bias Test
------------------------------------
t-value prob sig
Sign Bias 1.453 1.465e-01
Negative Sign Bias 4.634 3.922e-06 ***
Positive Sign Bias 0.199 8.423e-01
Joint Effect 23.560 3.086e-05 ***
Adjusted Pearson Goodness-of-Fit Test:
------------------------------------
group statistic p-value(g-1)
1 20 19.98 0.3956
2 30 25.29 0.6631
3 40 28.14 0.9014
4 50 39.02 0.8456
Elapsed time : 0.3038 plot(VALE.garch11.t.fit, which=9)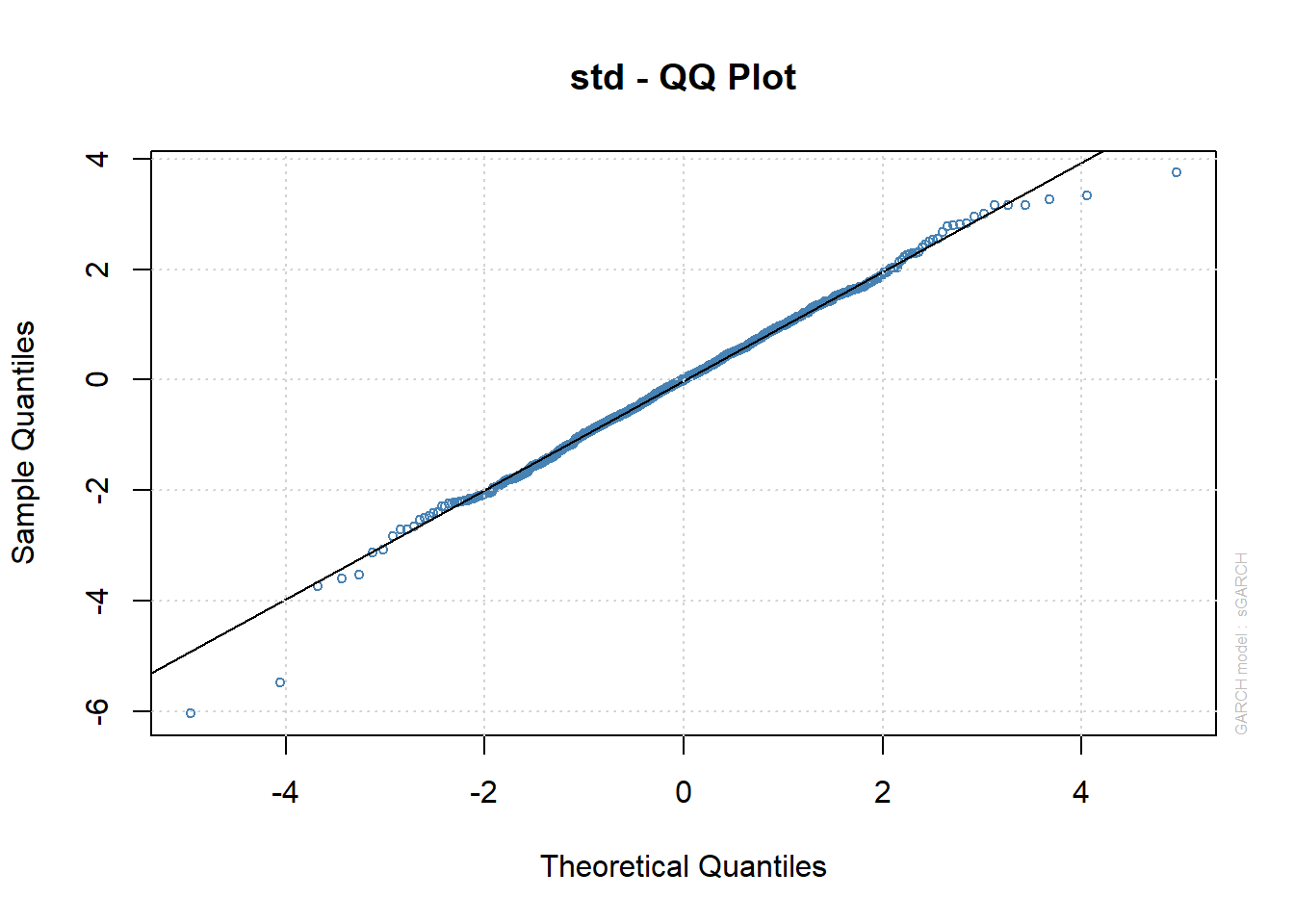
aparch11.1.t.spec = ugarchspec(variance.model = list(model="apARCH",
garchOrder=c(1,1)),
mean.model = list(armaOrder=c(0,0)),
distribution.model = "std",
fixed.pars=list(delta=1))
VALE.aparch11.1.t.fit = ugarchfit(spec=aparch11.1.t.spec, data=VALE.ret)
VALE.aparch11.1.t.fit
*---------------------------------*
* GARCH Model Fit *
*---------------------------------*
Conditional Variance Dynamics
-----------------------------------
GARCH Model : apARCH(1,1)
Mean Model : ARFIMA(0,0,0)
Distribution : std
Optimal Parameters
------------------------------------
Estimate Std. Error t value Pr(>|t|)
mu 0.000145 0.000647 0.22422 0.822584
omega 0.002495 0.000967 2.57914 0.009905
alpha1 0.082988 0.021633 3.83616 0.000125
beta1 0.842620 0.047924 17.58228 0.000000
gamma1 0.603759 0.192088 3.14313 0.001671
delta 1.000000 NA NA NA
shape 6.795048 1.197332 5.67516 0.000000
Robust Standard Errors:
Estimate Std. Error t value Pr(>|t|)
mu 0.000145 0.000655 0.22145 0.824740
omega 0.002495 0.001224 2.03781 0.041569
alpha1 0.082988 0.025608 3.24064 0.001193
beta1 0.842620 0.060979 13.81829 0.000000
gamma1 0.603759 0.180442 3.34600 0.000820
delta 1.000000 NA NA NA
shape 6.795048 1.198521 5.66953 0.000000
LogLikelihood : 3081
Information Criteria
------------------------------------
Akaike -4.4961
Bayes -4.4732
Shibata -4.4961
Hannan-Quinn -4.4875
Weighted Ljung-Box Test on Standardized Residuals
------------------------------------
statistic p-value
Lag[1] 0.003469 0.9530
Lag[2*(p+q)+(p+q)-1][2] 0.340440 0.7736
Lag[4*(p+q)+(p+q)-1][5] 1.592027 0.7173
d.o.f=0
H0 : No serial correlation
Weighted Ljung-Box Test on Standardized Squared Residuals
------------------------------------
statistic p-value
Lag[1] 18.62 1.591e-05
Lag[2*(p+q)+(p+q)-1][5] 19.00 3.644e-05
Lag[4*(p+q)+(p+q)-1][9] 20.07 1.924e-04
d.o.f=2
Weighted ARCH LM Tests
------------------------------------
Statistic Shape Scale P-Value
ARCH Lag[3] 0.1486 0.500 2.000 0.6999
ARCH Lag[5] 0.3035 1.440 1.667 0.9393
ARCH Lag[7] 0.9906 2.315 1.543 0.9151
Nyblom stability test
------------------------------------
Joint Statistic: 0.8841
Individual Statistics:
mu 0.07869
omega 0.16672
alpha1 0.17589
beta1 0.16925
gamma1 0.11133
shape 0.30942
Asymptotic Critical Values (10% 5% 1%)
Joint Statistic: 1.49 1.68 2.12
Individual Statistic: 0.35 0.47 0.75
Sign Bias Test
------------------------------------
t-value prob sig
Sign Bias 1.7339 8.317e-02 *
Negative Sign Bias 4.1472 3.574e-05 ***
Positive Sign Bias 0.1362 8.917e-01
Joint Effect 17.3662 5.942e-04 ***
Adjusted Pearson Goodness-of-Fit Test:
------------------------------------
group statistic p-value(g-1)
1 20 15.95 0.6608
2 30 29.41 0.4438
3 40 24.05 0.9711
4 50 38.14 0.8690
Elapsed time : 1.231 plot(residuals(VALE.aparch11.1.t.fit))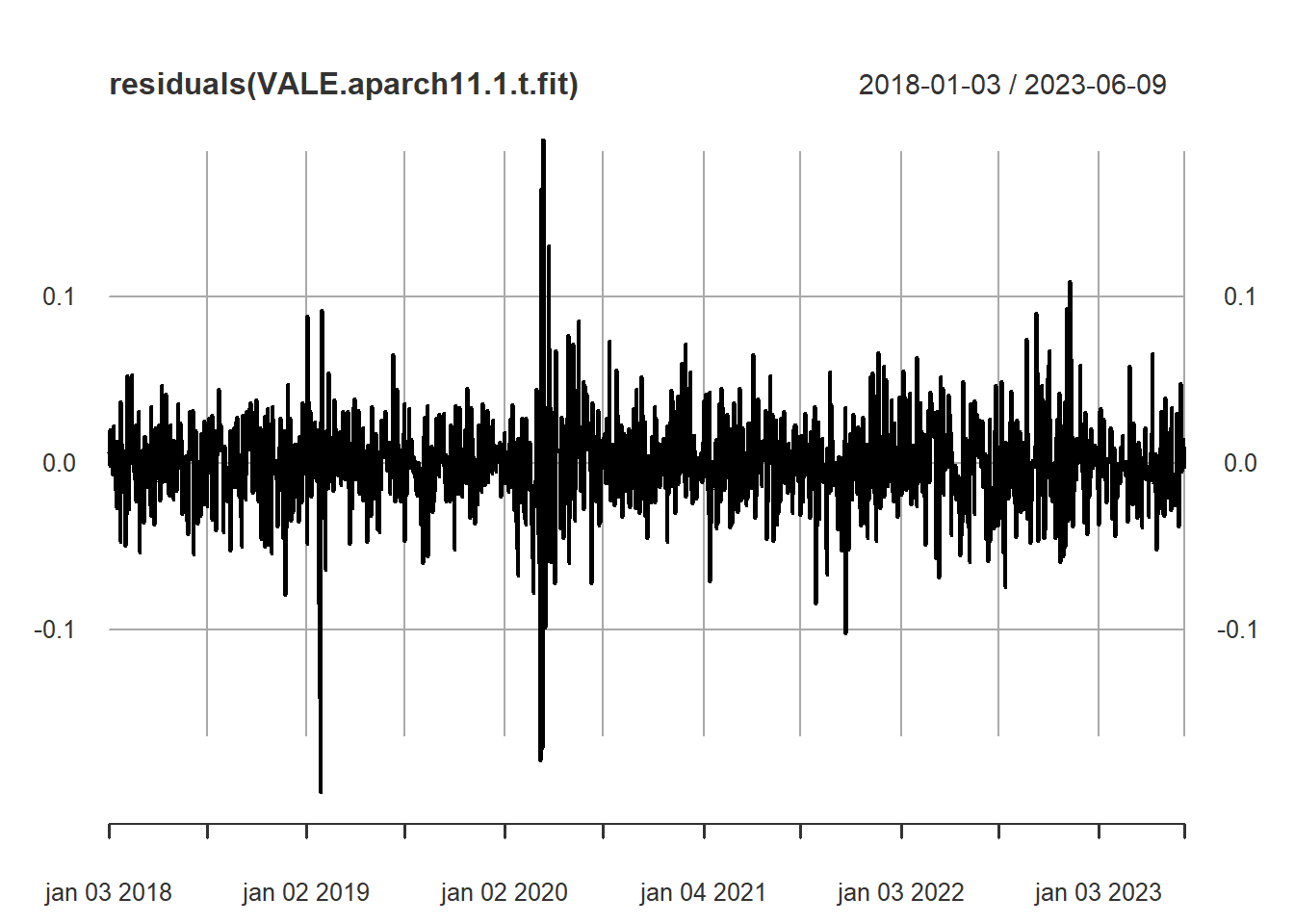
Ajustamos uma \(t\) enviesada:
garch11.st.spec = ugarchspec(variance.model = list(garchOrder=c(1,1)),
mean.model = list(armaOrder=c(0,0)),
distribution.model = "sstd")
VALE.garch11.st.fit = ugarchfit(spec=garch11.st.spec, data=VALE.ret)
VALE.garch11.st.fit
*---------------------------------*
* GARCH Model Fit *
*---------------------------------*
Conditional Variance Dynamics
-----------------------------------
GARCH Model : sGARCH(1,1)
Mean Model : ARFIMA(0,0,0)
Distribution : sstd
Optimal Parameters
------------------------------------
Estimate Std. Error t value Pr(>|t|)
mu 0.000340 0.000683 0.49827 0.618294
omega 0.000080 0.000032 2.51984 0.011741
alpha1 0.076986 0.022903 3.36144 0.000775
beta1 0.809743 0.059818 13.53670 0.000000
skew 0.971957 0.036344 26.74360 0.000000
shape 6.592137 1.135063 5.80773 0.000000
Robust Standard Errors:
Estimate Std. Error t value Pr(>|t|)
mu 0.000340 0.000673 0.5054 0.613279
omega 0.000080 0.000034 2.3344 0.019573
alpha1 0.076986 0.037494 2.0533 0.040042
beta1 0.809743 0.072072 11.2351 0.000000
skew 0.971957 0.037452 25.9523 0.000000
shape 6.592137 1.221276 5.3977 0.000000
LogLikelihood : 3076
Information Criteria
------------------------------------
Akaike -4.4876
Bayes -4.4647
Shibata -4.4877
Hannan-Quinn -4.4790
Weighted Ljung-Box Test on Standardized Residuals
------------------------------------
statistic p-value
Lag[1] 0.003709 0.9514
Lag[2*(p+q)+(p+q)-1][2] 0.279837 0.8070
Lag[4*(p+q)+(p+q)-1][5] 1.692357 0.6926
d.o.f=0
H0 : No serial correlation
Weighted Ljung-Box Test on Standardized Squared Residuals
------------------------------------
statistic p-value
Lag[1] 15.44 8.529e-05
Lag[2*(p+q)+(p+q)-1][5] 15.84 2.634e-04
Lag[4*(p+q)+(p+q)-1][9] 17.04 1.132e-03
d.o.f=2
Weighted ARCH LM Tests
------------------------------------
Statistic Shape Scale P-Value
ARCH Lag[3] 0.3944 0.500 2.000 0.530
ARCH Lag[5] 0.6753 1.440 1.667 0.831
ARCH Lag[7] 1.2759 2.315 1.543 0.865
Nyblom stability test
------------------------------------
Joint Statistic: 1.289
Individual Statistics:
mu 0.0969
omega 0.2857
alpha1 0.2098
beta1 0.3087
skew 0.5322
shape 0.3299
Asymptotic Critical Values (10% 5% 1%)
Joint Statistic: 1.49 1.68 2.12
Individual Statistic: 0.35 0.47 0.75
Sign Bias Test
------------------------------------
t-value prob sig
Sign Bias 1.4998 1.339e-01
Negative Sign Bias 4.6772 3.197e-06 ***
Positive Sign Bias 0.2336 8.153e-01
Joint Effect 23.9089 2.610e-05 ***
Adjusted Pearson Goodness-of-Fit Test:
------------------------------------
group statistic p-value(g-1)
1 20 15.95 0.6608
2 30 25.42 0.6562
3 40 28.32 0.8970
4 50 36.02 0.9162
Elapsed time : 0.4376 plot(VALE.garch11.st.fit, which=9)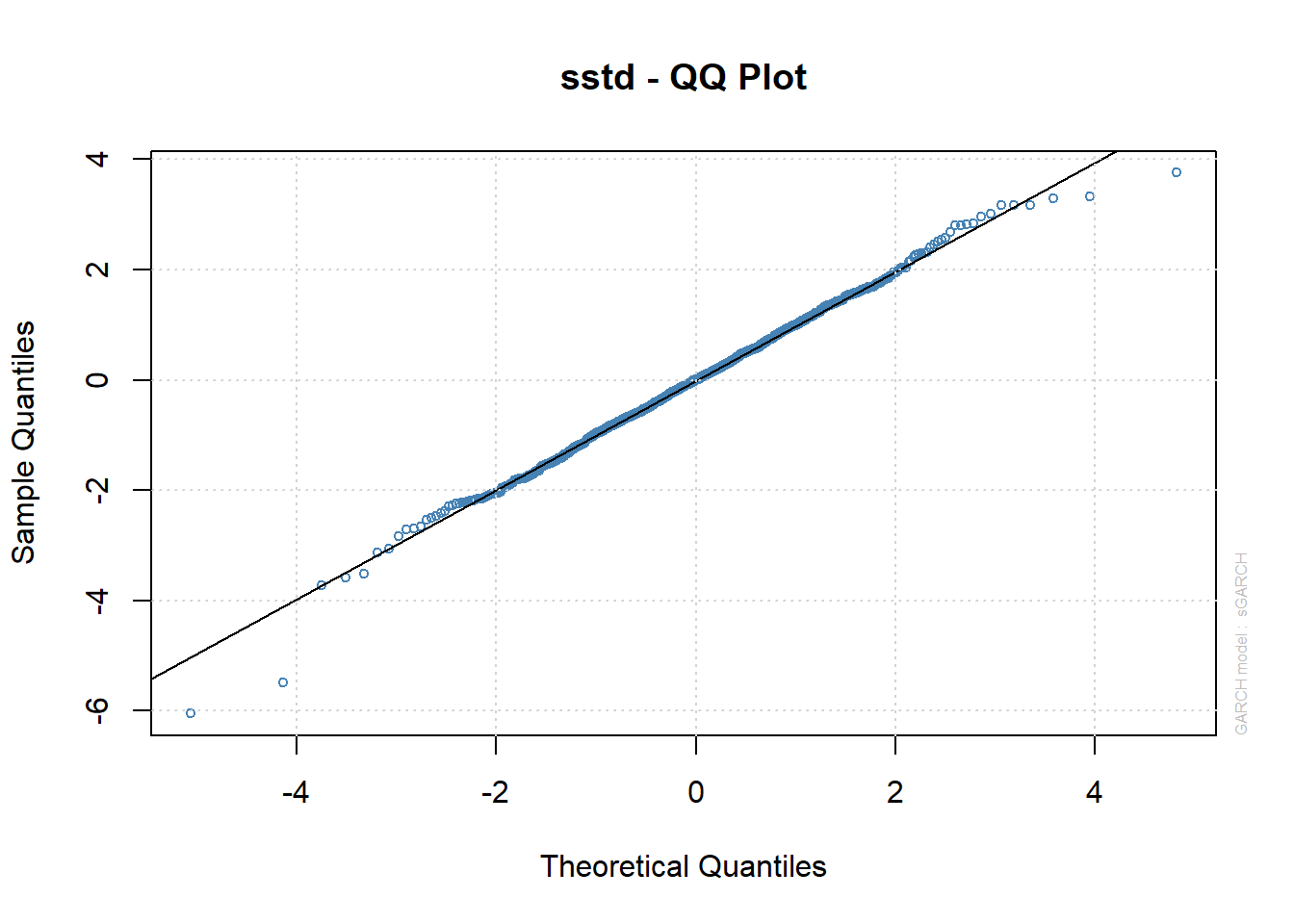
Plotamos os gráficos dos modelos da família ARCH
VALE.garch11.fcst = ugarchforecast(VALE.garch11.fit, n.ahead=250)
VALE.garch11.t.fcst = ugarchforecast(VALE.garch11.t.fit, n.ahead=250)
VALE.aparch11.1.fcst = ugarchforecast(VALE.aparch11.1.fit, n.ahead=250)
VALE.aparch11.1.t.fcst = ugarchforecast(VALE.aparch11.1.t.fit, n.ahead=250)Ploto os gráficos
par(mfrow=c(2,2))
plot(sigma(VALE.garch11.fcst), type="l", col="blue")
plot(sigma(VALE.garch11.t.fcst), type="l", col="blue")
plot(sigma(VALE.aparch11.1.fcst), type="l", col="blue")
plot(sigma(VALE.aparch11.1.t.fcst), type="l", col="blue")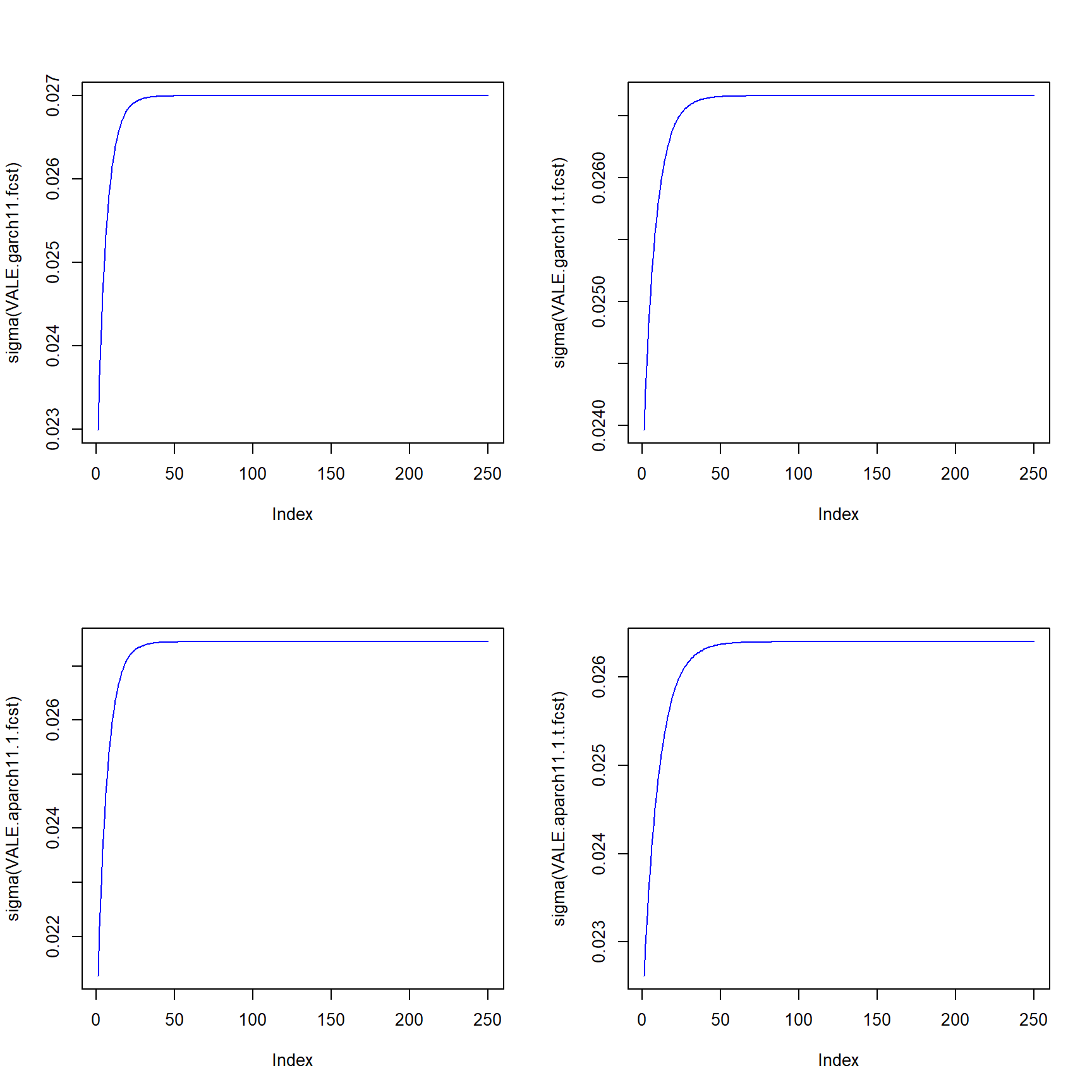
Extraio as projeções de volatilidades:
#VALE.garch11.sigma = as.data.frame(VALE.garch11.fcst)$sigma
#VALE.garch11.t.sigma = as.data.frame(VALE.garch11.t.fcst)$sigma
#VALE.aparch11.1.sigma = as.data.frame(VALE.aparch11.1.fcst)$sigma
#VALE.aparch11.1.t.sigma = as.data.frame(VALE.aparch11.1.t.fcst)$sigma
#ymax = max(VALE.garch11.sigma,VALE.garch11.t.sigma,VALE.aparch11.1.sigma, VALE.aparch11.1.t.sigma)
#ymin = min(VALE.garch11.sigma,VALE.garch11.t.sigma,VALE.aparch11.1.sigma, VALE.aparch11.1.t.sigma)
#plot.ts(VALE.garch11.sigma, main="Projeções de Volatilidades",
# ylim=c(ymin,ymax), col="black",
# lwd=2, ylab="sigma(t+h|t)", xlab="h")
#lines(VALE.garch11.t.sigma, col="blue", lwd=2)
#lines(VALE.aparch11.1.sigma, col="green", lwd=2)
#lines(VALE.aparch11.1.t.sigma, col="red", lwd=2)
#legend(x="topleft", legend=c("GARCH-n", "GARCH-t", "APARCH-n", "APARCH-t"),
# col=c("black", "blue","green","red"), lwd=2, lty = "solid")Reajusta os modelos, deixando 100 observações fora da amostra para estatísticas de avaliação de previsão
VALE.garch11.fit = ugarchfit(spec=garch11.spec, data=VALE.ret,
out.sample=100)
VALE.garch11.t.fit = ugarchfit(spec=garch11.t.spec, data=VALE.ret,
out.sample=100)
VALE.aparch11.1.fit = ugarchfit(aparch11.1.spec, VALE.ret,
out.sample=100)
VALE.aparch11.1.t.fit = ugarchfit(spec=aparch11.1.t.spec, data=VALE.ret,
out.sample=100)Compara a persistência com a variância incondicional:
c.mat = matrix(0, 4, 2)
colnames(c.mat) = c("Persistence", "E[sig(t)]")
rownames(c.mat) = c("GARCH-n", "GARCH-t", "APARCH-n","APARCH-t")
c.mat["GARCH-n","Persistence"] = persistence(VALE.garch11.fit)
c.mat["GARCH-t","Persistence"] = persistence(VALE.garch11.t.fit)
c.mat["APARCH-n","Persistence"] = persistence(VALE.aparch11.1.fit)
c.mat["APARCH-t","Persistence"] = persistence(VALE.aparch11.1.t.fit)
c.mat["GARCH-n","E[sig(t)]"] = sqrt(uncvariance(VALE.garch11.fit))
c.mat["GARCH-t","E[sig(t)]"] = sqrt(uncvariance(VALE.garch11.t.fit))
c.mat["APARCH-n","E[sig(t)]"] = sqrt(uncvariance(VALE.aparch11.1.fit))
c.mat["APARCH-t","E[sig(t)]"] = sqrt(uncvariance(VALE.aparch11.1.t.fit))
c.mat Persistence E[sig(t)]
GARCH-n 0.8402 0.02746
GARCH-t 0.8746 0.02709
APARCH-n 0.8522 0.02784
APARCH-t 0.8992 0.02672Finaliza computando 100 previsões dinâmicas 1 passo à frente:
VALE.garch11.fcst = ugarchforecast(VALE.garch11.fit, n.roll=100, n.ahead=1)
VALE.garch11.t.fcst = ugarchforecast(VALE.garch11.t.fit, n.roll=100, n.ahead=1)
VALE.aparch11.1.fcst = ugarchforecast(VALE.aparch11.1.fit, n.roll=100, n.ahead=1)
VALE.aparch11.1.t.fcst = ugarchforecast(VALE.aparch11.1.t.fit, n.roll=100, n.ahead=1)Vamos ver os resultados para os retornos esperados pelos 4 modelos ajustados:
VALE.garch11.fcst
*------------------------------------*
* GARCH Model Forecast *
*------------------------------------*
Model: sGARCH
Horizon: 1
Roll Steps: 100
Out of Sample: 1
0-roll forecast [T0=2023-01-17]:
Series Sigma
T+1 0.0008252 0.02441VALE.garch11.t.fcst
*------------------------------------*
* GARCH Model Forecast *
*------------------------------------*
Model: sGARCH
Horizon: 1
Roll Steps: 100
Out of Sample: 1
0-roll forecast [T0=2023-01-17]:
Series Sigma
T+1 0.0008341 0.02453VALE.aparch11.1.fcst
*------------------------------------*
* GARCH Model Forecast *
*------------------------------------*
Model: apARCH
Horizon: 1
Roll Steps: 100
Out of Sample: 1
0-roll forecast [T0=2023-01-17]:
Series Sigma
T+1 2.16e-05 0.02453VALE.aparch11.1.t.fcst
*------------------------------------*
* GARCH Model Forecast *
*------------------------------------*
Model: apARCH
Horizon: 1
Roll Steps: 100
Out of Sample: 1
0-roll forecast [T0=2023-01-17]:
Series Sigma
T+1 0.0004423 0.02365Com os dados observados, comparamos o de melhor ajuste:
# Obtem comparativo de previsao estatistica
fcst.list = list(garch11=VALE.garch11.fcst,
garch11.t=VALE.garch11.t.fcst,
aparch11.1=VALE.aparch11.1.fcst,
aparch11.t.1=VALE.aparch11.1.t.fcst)
fpm.mat = sapply(fcst.list, fpm)
fpm.mat garch11 garch11.t aparch11.1 aparch11.t.1
MSE 0.0004522 0.0004523 0.0004476 0.0004498
MAE 0.01625 0.01625 0.01612 0.01618
DAC 0.43 0.43 0.43 0.43 Referências
Graves, Spencer. 2014. FinTS: Companion to Tsay (2005) Analysis of Financial Time Series. https://CRAN.R-project.org/package=FinTS.
Ghalanos, Alexios. 2015. Rugarch: Univariate Garch Models. https://CRAN.R-project.org/package=rugarch.